抗性基因數據庫CARD介紹
隨著抗生素藥物的發現及使用,越來越多的耐藥菌株由此產生。而耐藥菌株的發展則會增加疾病治療的難度和成本,因此耐藥微生物的研究則顯得尤為重要。目前,通過對耐藥基因的鑒定挖掘能夠一定程度上幫助我們揭開耐藥機制,為疾病的治療、藥物研發提供參考。ARDB是最先整合了各種微生物中抗藥基因的數據庫,但它從2009年開始就不再更新。而CARD(the Comprehensive Antibiotic Research Database)數據庫包含了ARDB數據庫中所有抗性信息,並搭建了一個基於誌願者貢獻的數據共享平臺,做到了實時更新保證了數據的有效性。目前,CARD數據庫收集了超過1600個已知的抗生素抗性基因。
數據庫描述
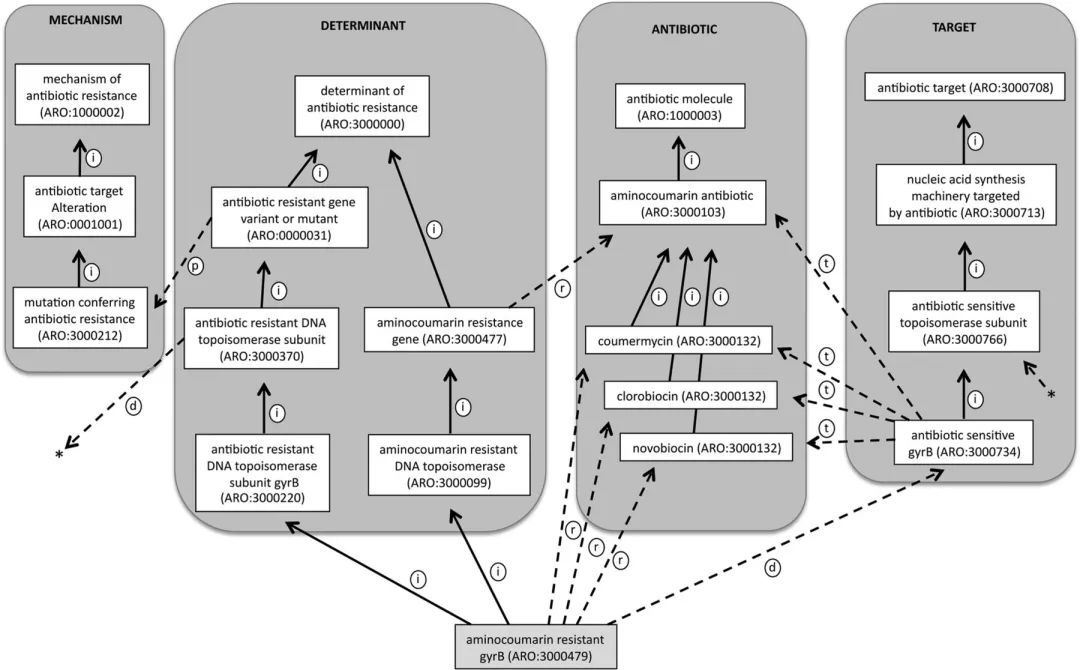
CARD數據庫(http://arpcard.mcmaster.ca)核心是ARO(Antibiotic Resistance Ontology), ARO包含了與抗生素抗性基因,抗性機制,抗生素和靶相關的term,如圖所示。2017年發表的文章中,更新了數據庫的相關功能,其中也提到了其他本體論,如用於描述抗生素抗性基因預測模塊和參數的MO,定義不同term之間關系類型的RO,以及描述CARD中物種和菌株的NCBITaxon。
耐藥基因預測
在CARD數據庫網站,點擊Analyze選項可進入耐藥基因預測界面。耐藥基因預測分析可通過選擇BLAST和RGI(Resistance Gene Identifier)兩種模式來實現。BLAST是依賴NCBI中BLAST軟件,將序列與CARD參考序列進行比對,獲得相關的註釋信息; RGI是CARD數據庫團隊開發的基於蛋白預測抗性基因序列的軟件,即通過蛋白同源和蛋白變異來預測抗性基因序列。目前,RGI僅能夠分析蛋白序列,如果有基因組序列或組裝後的contigs提交上來,那麽首先需要使用軟件Prodigal來預測開放閱讀框,然後RGI分析預測得到的蛋白序列。
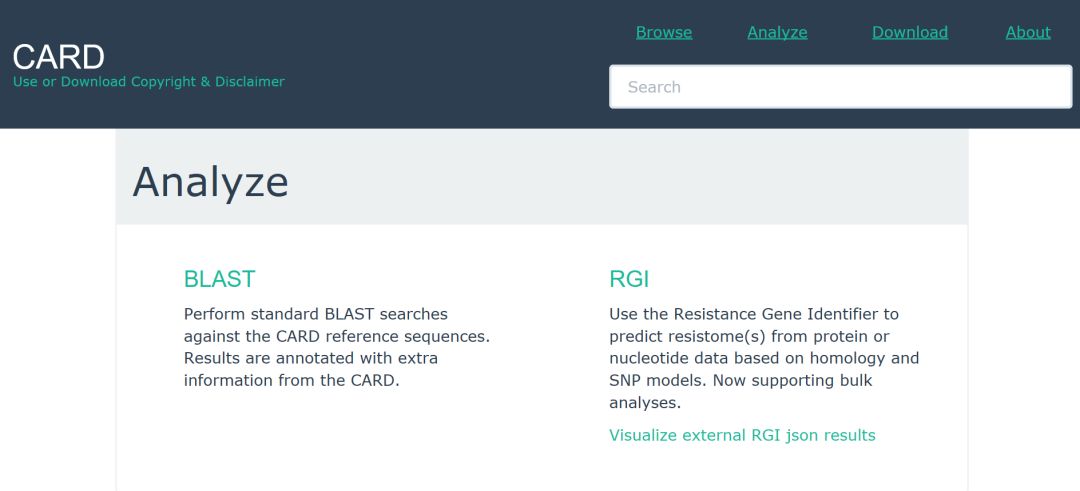
圖1
RGI接受多種格式的數據信息,包括: GenBank accession ,GI 號,fasta格式的序列信息等,見圖2。在進行耐藥基因預測時,提供了三種算法,即Perfect, Strict, and Loose。RGI一般默認Perfect, Strict。RGI結果可通過Resistance wheel可視化,見圖3。圖中內環表示抗性分類,外環代表抗生素抗性基因,例如抗性基因orf0_267表現氨基糖苷類(aminoglycoside)抗生素抗性。
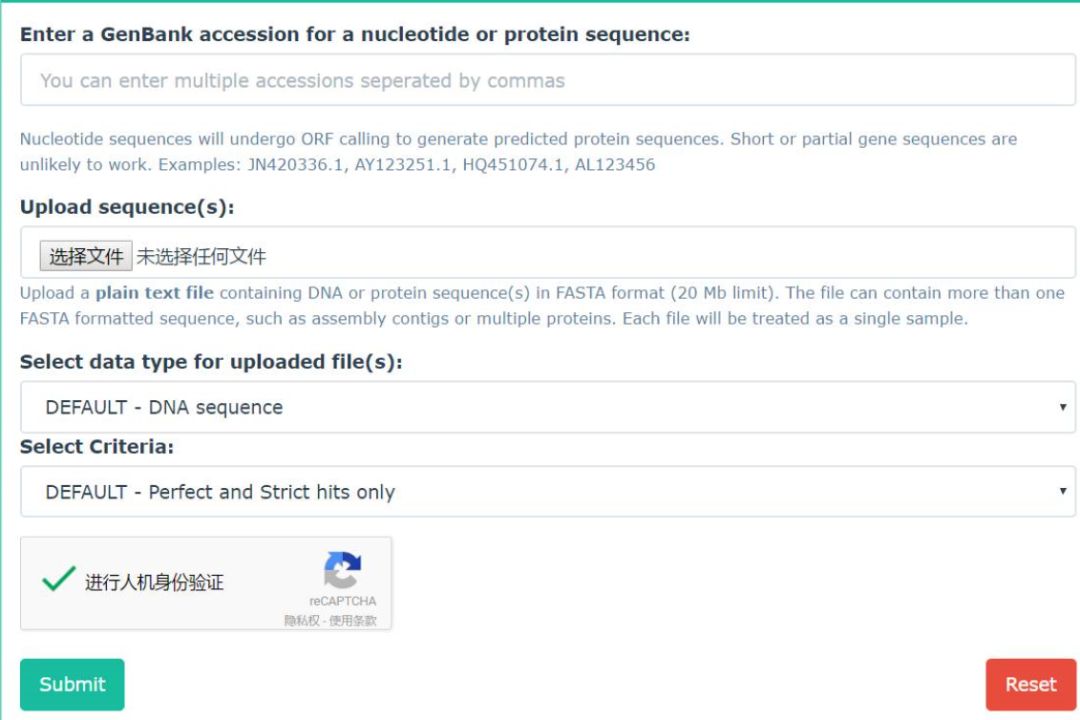
圖2
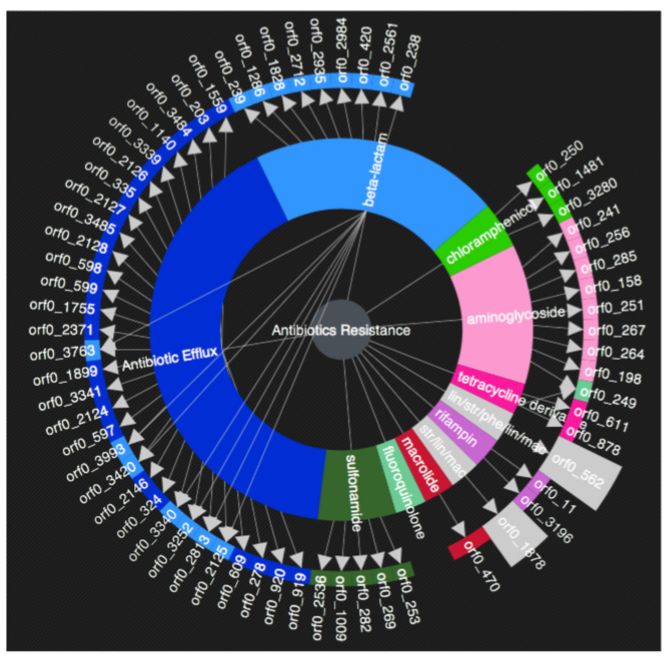
圖3
數據庫下載
所有的ARO,靶,序列等都能夠被下載,下載地址為http://arpcard.mcmaster.ca/download。如圖4所示,ARO以OBO格式存在,每個term都包含term id號,抗生素分類信息,抗生素描述信息等;序列數據以fasta格式存在,如圖5。

圖4 ARO
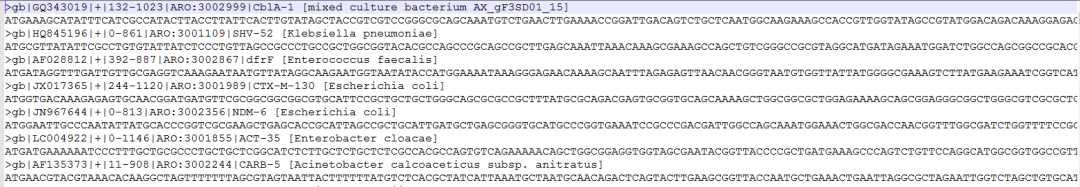
圖5 nucleotide_fasta_protein_homolog_model
CARD數據庫下載之後,可以通過blastp將基因序列比對到CARD數據庫,從而進行抗性基因的註釋。如果給出的是組裝後的scaffold序列,則需要先進行基因預測,然後將預測得到的蛋白序列比對到CARD數據庫,從而獲得抗性基因。
參考文獻:
McArthur,A.G., Waglechner,N., Nizam,F., Yan,A., Azad,M.A., Baylay,A.J., Bhullar,K., Canova,M.J., de Pascale,G., Ejim,L. et al. (2013) The comprehensive antibiotic resistance database. Antimicrob. Agents Chemother., 57, 3348–3357.
2. Jia B, Raphenya AR, Alcock B, Waglechner N, Guo P, Tsang KK, Lago BA, Dave BM, Pereira S, Sharma AN.et al. (2017) CARD 2017: expansion and model-centric curation of the comprehensive antibiotic resistance database.Nucleic acids research Volume 45 p.D566-D573
抗性基因數據庫CARD介紹
