遺傳演算法+BP神經網路組合求解非線性函式
用遺傳演算法求解非線性問題是常見的求解演算法之一,求解的過程就是隨機生成解,計算適應度,然後選擇,交叉,變異,更新種群,不斷迭代,這樣,每個個體都會向每代中最佳的個體學習並靠攏,這是區域性最優解;而變異操作是為了在靠近當前最優解的同時還有機會變異出更佳的基因,從而跳出區域性最優解而達到全域性最優解。
而有時候,面對一個很複雜的非線性函式,或者是根本無法用確定的表示式描述的離散非線性函式,在計算適應度時就會產生很大的問題,比如計算時間過長,解出式子需要半個小時;比如無法計算,只有貼近的離散點。這樣,傳統的遺傳演算法無法達到我們期望的速度和要求,我們就需要引入其他輔助遺傳演算法的內容。
這裡,我們引入了BP神經網路:有導師學習的誤差前饋神經網路。BP神經網路隨機初始化權值與閾值,並通過已有的訓練資料和訓練期望,將計算出來的誤差前饋給神經網路,也就是歸結為權值和閾值的“過錯”。這樣,權值和閾值不斷得到修改,最終形成逼近訓練資料和期望的模型。
所以,我們在遺傳演算法的主過程中,先迭代一部分的次數,得到一部分種群個體和適應度值,用來訓練BP神經網路。接下來的迭代中,將一部分的種群個體的適應度直接用BP模型求出來,而另一部分的種群個體適應度仍然用原函式求出(或者原來的預測曲線),將這一部分的種群個體和適應度再次帶入BP神經網路中訓練網路,使網路越來越精準。神經網路的求值是很快的,最後時間複雜度將會大大降低。
在下面的例子中,有一個很簡單的例子:。這是一個很簡單的非線性函式,我們用上面的思路來模擬一遍解法,最後得出最優解和一個逼近此函式的BP網路模型。
首先,遺傳演算法的基本函式如下
1.選擇函式,以每個個體的適應度為概率選擇優秀個體,更新種群select.m
function ret=select(individuals,sizepop) % 本函式對每一代種群中的染色體進行選擇,以進行後面的交叉和變異 % individuals input : 種群資訊 % sizepop input : 種群規模 % opts input : 選擇方法的選擇 % ret output : 經過選擇後的種群 individuals.fitness= 1./(individuals.fitness); sumfitness=sum(individuals.fitness); sumf=individuals.fitness./sumfitness; index=[]; for i=1:sizepop %轉sizepop次輪盤 pick=rand; while pick==0 pick=rand; end for j=1:sizepop pick=pick-sumf(j); if pick<0 index=[index j]; break; %尋找落入的區間,此次轉輪盤選中了染色體i,注意:在轉sizepop次輪盤的過程中,有可能會重複選擇某些染色體 end end end individuals.chrom=individuals.chrom(index,:); individuals.fitness=individuals.fitness(index); ret=individuals
2.交叉函式:Cross.m
function ret=Cross(pcross,lenchrom,chrom,sizepop,bound)
%本函式完成交叉操作
% pcorss input : 交叉概率
% lenchrom input : 染色體的長度
% chrom input : 染色體群
% sizepop input : 種群規模
% ret output : 交叉後的染色體
for i=1:sizepop
% 隨機選擇兩個染色體進行交叉
pick=rand(1,2);
while prod(pick)==0
pick=rand(1,2);
end
index=ceil(pick.*sizepop);
% 交叉概率決定是否進行交叉
pick=rand;
while pick==0
pick=rand;
end
if pick>pcross
continue;
end
flag=0;
while flag==0
% 隨機選擇交叉位置
pick=rand;
while pick==0
pick=rand;
end
pos=ceil(pick.*sum(lenchrom)); %隨機選擇進行交叉的位置,即選擇第幾個變數進行交叉,注意:兩個染色體交叉的位置相同
pick=rand; %交叉開始
v1=chrom(index(1),pos);
v2=chrom(index(2),pos);
chrom(index(1),pos)=pick*v2+(1-pick)*v1;
chrom(index(2),pos)=pick*v1+(1-pick)*v2; %交叉結束
flag1=test(lenchrom,bound,chrom(index(1),:)); %檢驗染色體1的可行性
flag2=test(lenchrom,bound,chrom(index(2),:)); %檢驗染色體2的可行性
if flag1*flag2==0
flag=0;
else flag=1;
end %如果兩個染色體不是都可行,則重新交叉
end
end
ret=chrom;
其中test函式為test.m,判斷是否合法
function flag=test(lenchrom,bound,code)
%判斷是否合法
% lenchrom input : 染色體長度
% bound input : 變數的取值範圍
% code output: 染色體的編碼值
flag=1;
[n,m]=size(code);
for i=1:n
if code(i)<bound(i,1) || code(i)>bound(i,2)
flag=0;
end
end3.變異函式:Mutation.m
function ret=Mutation(pmutation,lenchrom,chrom,sizepop,pop,bound)
% 本函式完成變異操作
% pcorss input : 變異概率
% lenchrom input : 染色體長度
% chrom input : 染色體群
% sizepop input : 種群規模
% pop input : 當前種群的進化代數和最大的進化代數資訊
% ret output : 變異後的染色體
for i=1:sizepop
% 隨機選擇一個染色體進行變異
pick=rand;
while pick==0
pick=rand;
end
index=ceil(pick*sizepop);
% 變異概率決定該輪迴圈是否進行變異
pick=rand;
if pick>pmutation
continue;
end
flag=0;
while flag==0
% 變異位置
pick=rand;
while pick==0
pick=rand;
end
pos=ceil(pick*sum(lenchrom)); %隨機選擇了染色體變異的位置,即選擇了第pos個變數進行變異
v=chrom(i,pos);
v1=v-bound(pos,1);
v2=bound(pos,2)-v;
pick=rand; %變異開始
if pick>0.5
delta=v2*(1-pick^((1-pop(1)/pop(2))^2));
chrom(i,pos)=v+delta;
else
delta=v1*(1-pick^((1-pop(1)/pop(2))^2));
chrom(i,pos)=v-delta;
end %變異結束
flag=test(lenchrom,bound,chrom(i,:)); %檢驗染色體的可行性
end
end
ret=chrom;4.main函式,就是我們的主過程,
先給出其中Code.m的程式碼:
function ret=Code(lenchrom,bound)
%本函式將變數編碼成染色體,用於隨機初始化一個種群
% lenchrom input : 染色體長度
% bound input : 變數的取值範圍
% ret output: 染色體的編碼值
flag=0;
while flag==0
pick=rand(1,length(lenchrom));
ret=bound(:,1)'+(bound(:,2)-bound(:,1))'.*pick; %線性插值
flag=test(lenchrom,bound,ret); %檢驗染色體的可行性
end然後,整個過程就出來了,還是很簡單的
main.m
clc
clear all
close all
%引數
maxgen=100; %迭代次數
sizepop=100; %種群規模
pcross=[0.6]; %交叉概率
pmutation=[0.01];
lenchrom=[1 1 1 1 1]; %個體長度
bound=[0 0.9*pi;0 0.9*pi;0 0.9*pi;0 0.9*pi;0 0.9*pi];%五個引數都是一樣的域
%個體結構體
individuals=struct('fitness',zeros(1,sizepop),'chrom',[]);
avgfitness=[]; %平均適應度
bestfitness=[]; %最佳適應度
bestchrom=[]; %最佳種群個體
Testfitness=[];
Testchrom=[];
%初始化種群
for i=1:sizepop
%隨機產生種群
individuals.chrom(i,:)=Code(lenchrom,bound);
x=individuals.chrom(i,:);
individuals.fitness(i)=fun(x);
end
%找最好的染色體
[n_bestfitness,bestindex]=min(individuals.fitness);
%最好的染色體
bestchrom=individuals.chrom(bestindex,:);
avgfitness=sum(individuals.fitness)/sizepop; %平均適應度
trace=[];%每一代最好的適應度
%先進化二十代,選出20*100個個體和適應度,並訓練神經網路
for i=1:20
%選擇
individuals=select(individuals,sizepop);
avgfitness=sum(individuals.fitness)/sizepop;
%交叉
individuals.chrom=Cross(pcross,lenchrom,individuals.chrom,sizepop,bound);
%變異
individuals.chrom=Mutation(pmutation,lenchrom,individuals.chrom,sizepop,[i maxgen],bound);
%計算適應度
for j=1:sizepop
x=individuals.chrom(j,:);%一個個體
individuals.fitness(j)=fun(x);
Testfitness=[Testfitness;fun(x)]; %將每個個體都加入訓練矩陣中,擴大訓練樣本
Testchrom=[Testchrom;x];
end
%找到最好的染色體和他們在種群的位置
[newbestfitness newbestindex]=min(individuals.fitness);
[worestfitness,worestindex]=max(individuals.fitness);
if n_bestfitness>newbestfitness
bestindex=newbestindex;
bestchrom=individuals.chrom(bestindex,:);
n_bestfitness=individuals.fitness(bestindex);
end
individuals.chrom(worestindex,:)=bestchrom;
individuals.fitness(worestindex)=n_bestfitness;
avgfitness=sum(individuals.fitness)/sizepop;
trace=[trace;avgfitness n_bestfitness];
end
%%新建bp神經網路
P=Testchrom';
T=Testfitness';
inputnum=size(P,1);
outputnum=size(T,1);
hiddennum=11;
net=newff(Testchrom',Testfitness',hiddennum);
%設定訓練引數
net.trainParam.epochs=1000;
net.trainParam.goal=0.01;
LP.lr=0.1;
net.trainParam.show=NaN;
%訓練bp網路
net=train(net,P,T);
%訓練好之後,繼續進行遺傳的迭代,不過這次計算適應度一半用遺傳,一半用bp,並且遺傳的一部分繼續用於訓練bp
for i=21:maxgen
%選擇
individuals=select(individuals,sizepop);
avgfitness=sum(individuals.fitness)/sizepop;
%交叉
individuals.chrom=Cross(pcross,lenchrom,individuals.chrom,sizepop,bound);
%變異
individuals.chrom=Mutation(pmutation,lenchrom,individuals.chrom,sizepop,[i maxgen],bound);
%計算適應度
is_bpfun=[];
mid=50; %可以自動設定比例,決定哪一半用BP計算
for j=1:sizepop
if j<mid
%前mid個個體用直接計算的方法
x=individuals.chrom(j,:);
individuals.fitness(j)=fun(x);
Testfitness=[Testfitness;individuals.fitness(j)];
is_bpfun(:,j)=0;
Testchrom=[Testchrom;x];
else
if j==mid
%第mid代就可以訓練網路
x=individuals.chrom(j,:);
individuals.fitness(j)=fun(x);
is_bpfun(:,j)=0;
Testfitness=[Testfitness;individuals.fitness(j)];
Testchrom=[Testchrom;x];
net.train(net,Testchrom',Testfitness');
else
%大於mid的用神經網路直接輸出結果
x=individuals.chrom(j,:);
individuals.fitness(j)=sim(net,x');
is_bpfun(:,j)=1;
end
end
end
[newbestfitness,newbestindex]=min(individuals.fitness);
[worestfitness,worestindex]=max(individuals.fitness);
if n_bestfitness>newbestfitness
bestindex=newbestindex;
bestchrom=individuals.chrom(bestindex,:);
n_bestfitness=individuals.fitness(bestindex);
end
%如果是神經網路算出的結果,重算
while is_bpfun(bestindex)==1
x=individuals.chrom(bestindex,:);
individuals.fitness(bestindex)=fun(x);
if individuals.fitness(bestindex)>n_bestfitness
is_bpfun(bestindex)=0;
n_bestfitness=individuals.fitness(bestindex);
else
[newbestfitness newbestindex]=min(individuals.fitness);
bestindex=newbestindex;
n_bestfitness=newbestfitness;
end
end
individuals.chrom(worestindex,:)=bestchrom;
individuals.fitness(worestindex)=n_bestfitness;
avgfitness=sum(individuals.fitness)/sizepop;
trace=[trace;avgfitness n_bestfitness];
end
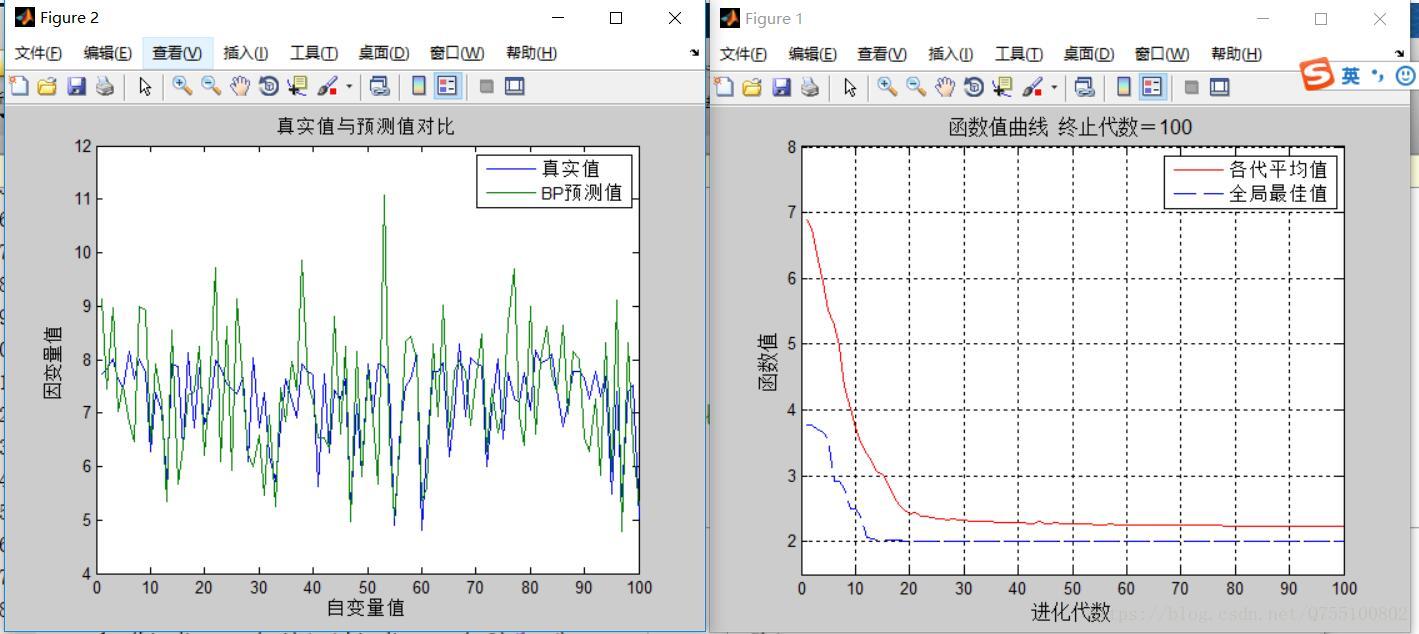
%% 結果顯示
[r c]=size(trace);
figure
plot([1:r]',trace(:,1),'r-',[1:r]',trace(:,2),'b--');
title(['函式值曲線 ' '終止代數=' num2str(maxgen)],'fontsize',12);
xlabel('進化代數','fontsize',12);ylabel('函式值','fontsize',12);
legend('各代平均值','全域性最佳值','fontsize',12);
disp('函式值 變數');
ylim([1.5 8])
%xlim([1,size(trace,1)])
grid on
% 視窗顯示
disp([n_bestfitness x]);
testchrom=[];
T_test=[];
%隨機生成測試資料
for i=1:sizepop
testchrom(i,:)=Code(lenchrom,bound);
x=testchrom(i,:);
T_test(i,:)=fun(x);
end
P_test=sim(net,testchrom');
P_test2=P_test';
N=0.9*pi;
x=0:N/20:N;
figure
plot([T_test P_test2]);
legend('真實值','BP預測值');
xlabel('自變數值');
ylabel('因變數值');
string={'真實值與預測值對比'};
title(string);
最後,執行程式,得到的結果如下圖