REST easy with kbmMW #17 – Database 6 – Existing databases
kbmMW已經包含了非常精細的功能來確定和解釋資料庫中表的元資料。
在下一版本中,這個功能將得到進一步加強,可以匯入現有資料庫中的表並自動建立與表相匹配的ORM類。
這意味著你能夠使用kbmMW的ORM智慧來查詢表,將資料儲存回表並操縱表元資料,例如新增,更改或刪除欄位和索引以及需要能夠升級資料庫用於匹配已更改的ORM表類的更高版本的表。
當你意識到需要更改資料庫表以允許其他或不同的欄位時,ORM的設計目標之一始終是抽象掉所需的所有枯燥工作。在kbmMW中,它就像更改ORM類一樣簡單,並使用kbmMW的ORM來建立表的CreateOrUpgrade。這已經在早期的REST相關文章中介紹過。
包含在下一個即將釋出的kbmMW版本中的是ORM本身為Delphi類發出程式碼的能力,該程式碼與ORM已經瞭解的任何表匹配。由於ORM已經支援分析現有的表結構以確定現有的ORM類是否與實際的表結構不同,為了能夠自動升級它,它是下一個邏輯步驟,能夠使用有關表結構的知識和元資料生成全新的資料庫表Delphi類。
這已被實現到一個簡單的工具:ImportDBSchema,現在我來演示這個工具的用法:
該工具的程式碼實際上非常簡單,因為複雜的部分包含在kbmMW的內部。
基本上它使用kbmMW和FireDAC連線到kbmMW的ORM和FireDAC支援的任何資料庫,它們是SQLite,MSSQL,MySQL,Oracle,Interbase / Firedac和PostgreSQL,並從連線的資料庫中選擇一個或多個表,之後那些表格“浮出水面”到kbmMW並生成程式碼。
表格表示kbmMW語言,從表中建立足夠的元資料資訊,以便能夠使用kbmMW的ORM註冊TkbmMWORMTable,基本上啟用查詢等,而無需實際擁有ORM類。
但由於TkbmMWORMTable包含kbmMW的ORM執行所需的所有元資料,因此它也可以用作生成新ORM表類的源。ORM類顯然使您可以非常輕鬆地將表的欄位和記錄作為具有屬性的常規物件進行訪問。
事實上,匯入包括生成ORM類的表,歸結為呼叫這兩個ORM方法:
var tbl:TkbmMWORMTable; begin ... tbl:=MyORM.SurfaceDynamicTable('actualdbtablename',true); MyORM.GenerateDelphiClass(tbl...); ... end;
讓我們嘗試從Ensembl基因組MySQL資料庫匯入一個表。
在這種情況下,我們使用匿名使用者名稱在伺服器ensembldb.ensembl.org上選擇名為aedes_aegypti_core_48_1b的資料庫。
可以在此頁面上找到連線資訊:https://www.ensembl.org/info/data/mysql.html
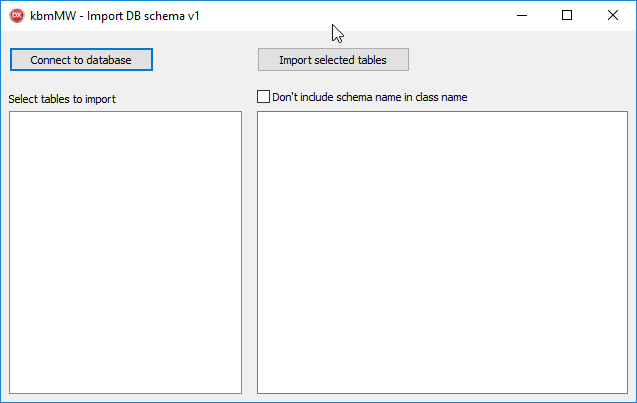
單擊“連線到資料庫”將開啟FireDAC對話方塊,我們可以在其中指定與資料庫的連線:

單擊“確定”,將顯示該資料庫(或模式)中的表列表,並選擇其中一些表進行匯入,然後單擊“匯入所選表”。
現在,系統會提示我們輸入用於儲存生成的類的檔案的單元檔名。
由於我們選擇了兩個表,因此該單元將包含兩個類。單擊“確定”後,將開始匯入。可能需要一段時間。
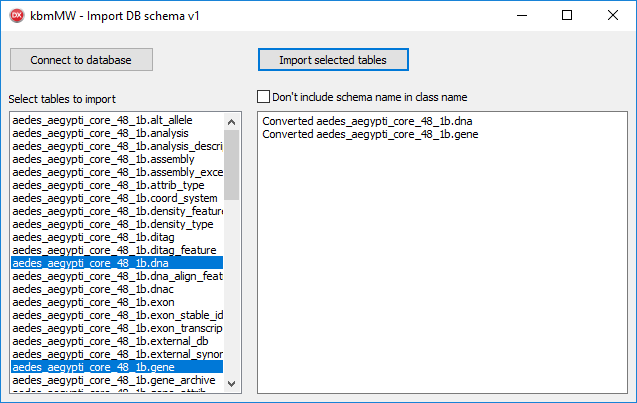
讓我們看看生成的單元,我稱之為ensembl.pas
unit ensembl; interface uses DB, Math, Classes, System.Generics.Collections, kbmMWGlobal, kbmMWObjectMarshal, kbmMWNullable, kbmMWORM, kbmMWRTTI; type [kbmMW_Table('name:aedes_aegypti_core_48_1b.dna')] TAedes_aegypti_core_48_1b_dna = class private FSeq_region_id:integer; FSequence:string; public [kbmMW_Field('name:seq_region_id, primary:true', ftInteger)] [kbmMW_NotNull] property Seq_region_id:integer read FSeq_region_id write FSeq_region_id; [kbmMW_Field('name:sequence', ftWideMemo)] [kbmMW_NotNull] property Sequence:string read FSequence write FSequence; end; [kbmMW_Table('name:aedes_aegypti_core_48_1b.gene')] TAedes_aegypti_core_48_1b_gene = class private FGene_id:integer; FBiotype:string; FAnalysis_id:word; FSeq_region_id:integer; FSeq_region_start:integer; FSeq_region_end:integer; FSeq_region_strand:byte; FDisplay_xref_id:kbmMWNullable<integer>; FSource:string; FStatus:kbmMWNullable<string>; FDescription:kbmMWNullable<string>; FIs_current:byte; public [kbmMW_Field('name:gene_id, primary:true', ftInteger)] [kbmMW_NotNull] property Gene_id:integer read FGene_id write FGene_id; [kbmMW_Field('name:biotype', ftString, 40)] [kbmMW_NotNull] property Biotype:string read FBiotype write FBiotype; [kbmMW_Field('name:analysis_id', ftSmallInt)] [kbmMW_NotNull] property Analysis_id:word read FAnalysis_id write FAnalysis_id; [kbmMW_Field('name:seq_region_id', ftInteger)] [kbmMW_NotNull] property Seq_region_id:integer read FSeq_region_id write FSeq_region_id; [kbmMW_Field('name:seq_region_start', ftInteger)] [kbmMW_NotNull] property Seq_region_start:integer read FSeq_region_start write FSeq_region_start; [kbmMW_Field('name:seq_region_end', ftInteger)] [kbmMW_NotNull] property Seq_region_end:integer read FSeq_region_end write FSeq_region_end; [kbmMW_Field('name:seq_region_strand', ftShortInt)] [kbmMW_NotNull] property Seq_region_strand:byte read FSeq_region_strand write FSeq_region_strand; [kbmMW_Field('name:display_xref_id', ftInteger)] property Display_xref_id:kbmMWNullable<integer> read FDisplay_xref_id write FDisplay_xref_id; [kbmMW_Field('name:source', ftString, 20)] [kbmMW_NotNull] property Source:string read FSource write FSource; [kbmMW_Field('name:status', ftWideString, 19)] property Status:kbmMWNullable<string> read FStatus write FStatus; [kbmMW_Field('name:description', ftWideMemo)] property Description:kbmMWNullable<string> read FDescription write FDescription; [kbmMW_Field('name:is_current', ftShortInt)] [kbmMW_NotNull] property Is_current:byte read FIs_current write FIs_current; end; implementation initialization TkbmMWRTTI.EnableRTTI([TAedes_aegypti_core_48_1b_dna, TObjectList<TAedes_aegypti_core_48_1b_dna>, TAedes_aegypti_core_48_1b_gene, TObjectList<TAedes_aegypti_core_48_1b_gene>]); kbmMWRegisterKnownClasses([TAedes_aegypti_core_48_1b_dna, TObjectList<TAedes_aegypti_core_48_1b_dna>, TAedes_aegypti_core_48_1b_gene, TObjectList<TAedes_aegypti_core_48_1b_gene>]); end.
您可能會注意到,每個類名都包含架構/資料庫名稱和表名稱。如果您只想將表名作為類名的一部分,請在匯入之前選中“不要在類名中包含模式名稱”複選框。
在一些相當罕見的情況下,資料庫表可能已經定義了某些kbmMW未知的資料型別。在這種情況下,匯入仍將發生,但有問題的欄位將使用UNKNOWN資料型別進行標記。
kbmMW還嘗試確定其他索引和音序器資訊,並生成與之匹配的相關屬性。然而,如果推斷這些型別的資訊失敗,那麼它將被忽略優先順序,它將被忽略,因此繼續生成基本的ORM表類。
現在,通過將此單元新增到您的應用程式,並設定MySQL kbmMW連線池以指向我們用於匯入的相同(或類似)伺服器,我們現在可以使用ORM輕鬆地從這兩個表中查詢資料。
var lst:TObjectList<TAedes_aegypti_core_48_1b_dna>; begin lst:=MyORM.QueryList<TAedes_aegypti_core_48_1b_dna>([2000],mwoqoGT); ... end;
在上面的示例中,我們將獲得大於2000的主鍵的TAedes_aegypti_core_48_1b_dna物件列表。
如果對列表中的任何物件進行了任何更改,則可以通過以下方式輕鬆將更改解析回資料庫
MyORM.Persist(lst);
使用ORM,使用這兩個表的正確結構建立我們自己的資料庫也非常容易。資料庫甚至不必是基於MySQL的資料庫。它可能是kbmMW的ORM支援的任何一個。
MyOtherORM.CreateOrUpgradeTable([TAedes_aegypti_core_48_1b_dna,TAedes_aegypti_core_48_1b_gene])
關於如何以一種新的現代面向物件方式輕鬆地重用現有資料庫,這就結束了這一點。
如果您喜歡我所展示的內容,請通過分享我們的部落格和藝術品來傳播關於kbmMW的資訊。
https://components4developers.blog/2018/11/15/rest-easy-with-kbmmw-17-database-6/
