chip-seq技術
1.ChIP-seq簡介
染色質免疫共沉澱技術(Chromatin-immunoprecipitation)也稱結合位點分析法,被用於蛋白質與DNA的互動作用。該技術將染色質免疫沉澱與大規模並行DNA測序相結合起來鑑定與DNA相關蛋白結合部位。其可被用於精確繪製任意目的蛋白在全基因組上的結合位點。
主要原理是基於RNA分子與RNA結合蛋白在紫外照射下發生藕聯,以RNA結合蛋白的特異性抗體將RNA-蛋白質複合沉澱之後,回收其中的RNA片段,經過加接頭、RT-PCR等步驟,對這些分子進行高通量測序。
2.一般生物資訊分析流程為:
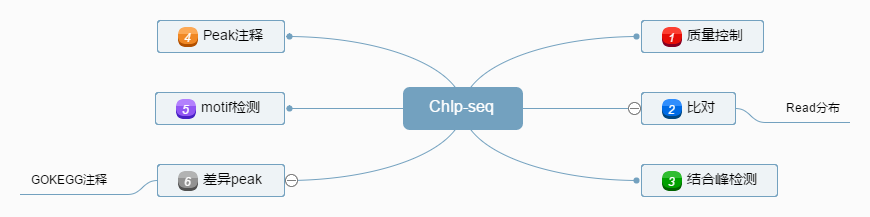
2.1 質量控制
2.2 比對
2.3 結合峰檢測
結合峰(peak)是通過演算法識別的富集區域,即識別的蛋白結合位點。將測序資料與參考基因組進行比對,
然後使用軟體對匹配到基因組上的短序列進行富集區域的掃描。通常,掃描到的富集區即被認為是蛋
白質與DNA 相互結合的區域。用於結合峰檢測的軟體最常用的有MACS2和Piranha。
軟體:Piranha,參考文獻: Site identification in high-throughput rna–protein interaction data.
軟體:MACS2,參考文獻:Model-based analysis of ChIP-Seq (MACS).
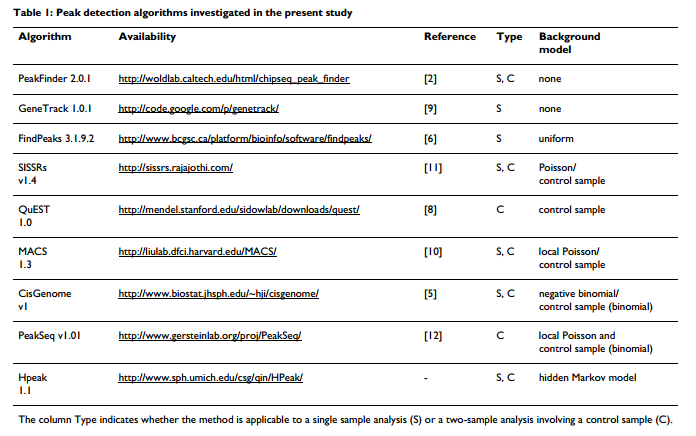
軟體比對參考文獻: A practical comparison of methods for detecting transcription factor binding sites in chip-seq experiments
2.4 結合峰的註釋
結合峰的註釋是指將peak註釋到所對應的基因資訊。
軟體:Homer,參考文獻:Simple combinations of lineage-determining transcription factors primecis-regulatory elements required for macrophage and B cell identities
其他資料:http://www.mi.fu-berlin.de/wiki/pub/ABI/GenomicsLecture14Materials/chipseq.pdf
https://bioinformatics-core-shared-training.github.io/cruk-autumn-school-2017/ChIP/Materials/Lectures/Lecture5_Peak%20Calling_SS.pdf
