heatmap繪製熱圖時出現樣本列名順序調換
阿新 • • 發佈:2019-01-02
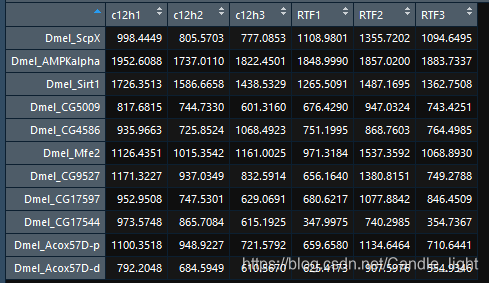
今天像往常一樣,用 ComplexHeatmap包中的 Heatmap函式畫熱圖,本來輸入是正常的
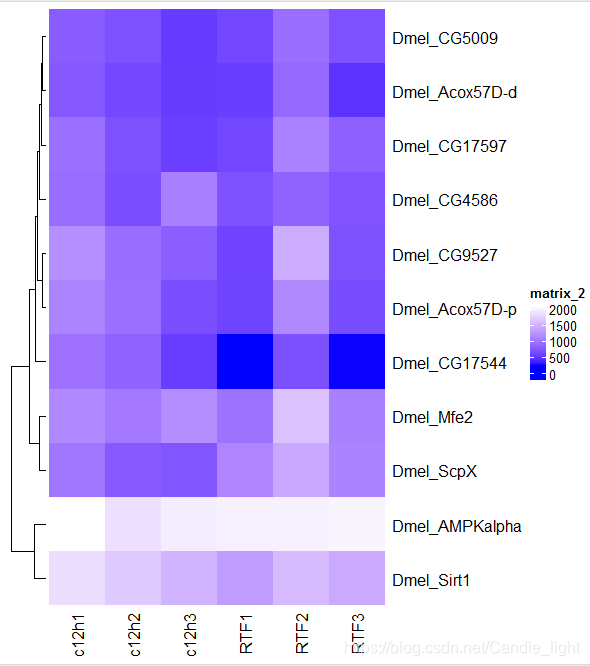
如圖,C12H在左,RTF在右
程式碼也沒啥問題
setwd("F:\\學習檔案存放地址\\IOZ_實驗室\\yangby\\結果檔案\\all_表達矩陣\\") library(ComplexHeatmap) library(circlize) #含註釋_all_deseq_CH_vs_RTF.csvtargetgene fil<-read.csv("含註釋_all_deseq_CH_vs_RTF.csvtargetgene.csv") paint_map_data<-fil row.names(paint_map_data)<-as.vector(fil[,16]) #設定圖的rowname paint_map_data<-paint_map_data[,c(10,11,12,13,14,15)] colnames(paint_map_data)<-c("c12h1","c12h2","c12h3","RTF1","RTF2","RTF3") #設定圖的colname Heatmap(paint_map_data[1:11,],c("blue","white")) #呼叫Heatmap函式展示基因表達量的熱圖
但是畫出來,竟然是這樣的?! 樣本列名順序不一致啦!
可以明顯看到樣本c12h3跑到了圖的左邊,RTF樣本名稱之間的順序也發生了變化,明明是RTF1,RTF2,RTF3排列的,現在變成了RTF1,RTF3,RTF2
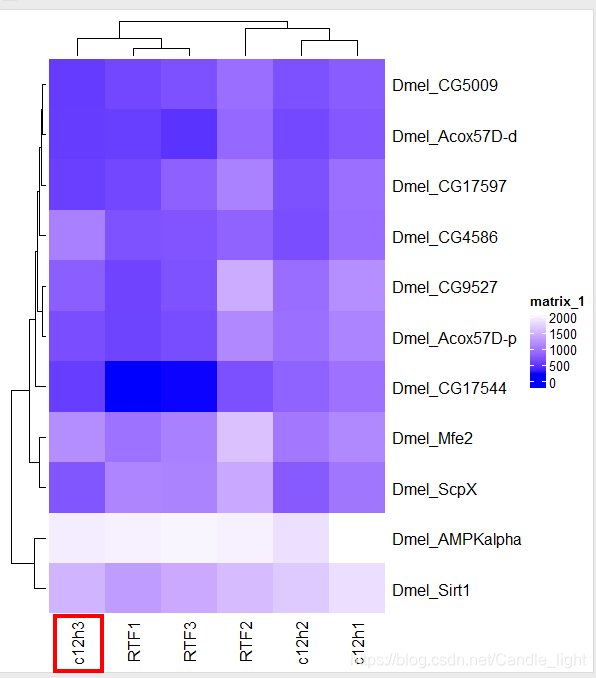
查看了一下Heatmap函式的引數,才知道原來這是由於對樣本的列進行了聚類導致的
因為聚類,將相似的樣本就放在一起了,所以樣本順序發生了變化,我們可以看到熱圖上方的聚類樹,這就是聚類的情況

解決辦法:畫圖的時候關掉對列樣本進行聚類的功能,如下設定:
Heatmap函式中新增引數**cluster_columns=F** 即可
Heatmap(paint_map_data[1:11,],c("blue","white"),cluster_columns=F) #調
大功告成: