風險預測模型評價第二彈:NRI的R語言計算
作者:麥子
轉載請註明:解螺旋·臨床醫生科研成長平臺
上期我們介紹了一個診斷或風險預測模型的評價指標,重新分類指數(Net reclassification index)。主要介紹了一些概念和運用,今天我們就來解決一下技術問題,怎麼算。想來想去,我覺得計算這個東西還是R比較威武。
話說恰好前兩天在知乎上看到某位生物學大大發了這麼一張圖——

啊呀啊喲!不服啊不服哎!可是想來好像又好有道理^(00)^
今天是R語言程式碼的盛(暴)宴(擊),除了NRI的運算,還有蠻多預處理的操作,在哪都能用得著。大家做好戰鬥準備。
R裡有2個包專事計算NRI,分別為nricens和PredictABEL。從最後結果來說,nricens計算出來的是絕對NRI,PredictABEL則為相對NRI。但我們已經知道計算原理了呀,而且它們都能生成新舊模型分類的對照表,所以其實只用其中一個包就都可以計算了。
不過它們還是有一些小小差異,我們就以logistic迴歸模型為例,分別看一下這兩個包,供大家參考選擇。Cox模型引數較多也較複雜,但我相信你看完這篇的講解就能看懂幫助文件中的cox案例,算是留個小作業給你吧~
擬合模型
先用一份示例資料做個模型。這是survival包裡帶有的一份梅奧診所的資料,記錄了418位患者的臨床指標,觀察這些因素與原發性膽汁性肝硬化(PBC)的關係。其中前312位參加了RCT,其他的只參加了觀察佇列。
我們用前312份樣本,做個預測2000天時間點上死亡與否的模型。先載入這份資料看一下。
library(survival)
### logistic迴歸
egData <- pbc[1:312,]

(點選看大圖)
做一個logistic迴歸,我們需要一個結局事件作為因變數,它必需是個分類變數;其次需要若干自變數,它們可以是分類也可以是連續。
這個表中的結局是status,0 = 截尾(刪失),1 = 接受移植,2 = 死亡。研究目的“死亡與否”是個二分類變數,所以要做些變換。
再看time一欄,有的不夠2000天,這些樣本要麼是沒到2000天就死亡了,要麼是刪失了。我們要刪掉2000天內刪失的資料。
egData = egData[egData$time > 2000 |
(egData$time < 2000 & egData$status == 2),]
“[ ]” 表示篩選條件,| 表示“或”,& 為“和”。所以條件句就是egData中的time一列大於2000的保留,或小於2000但同時狀態為死亡的也保留。最後一個“,”別忘了,其在條件句的前面表示對列進行選擇,在其後表示選擇行。
選好後做一個event向量,把status的三個狀態變成死亡 = 1, 未死亡 = 0。
event = ifelse(egData$time < 2000 & egData$status == 2, 1, 0)
ifelse (test, yes, no)大法好啊,前面一個test是邏輯判斷句,其值為真時返回yes的值,為假時返回no的值。所以本句中test就是當time<2000,且status為2(死亡)時,記為1,否則為0。
然後把event合併入原來的表格。
egData = cbind(egData,event)
cbind()是以列合併,另有rbind()以行合併。這樣event就成了最後一列,為結局事件。
然後選擇模型的自變數(predictors)。太多了,選取其中幾個示例。就以年齡、膽紅素、白蛋白為舊模型(standard),三者加上一個凝血酶原時間為新模型(new)。
一般做logistic迴歸是用glm(因變數 ~ 自變數1 + 自變數2 + …… +自變數n,family = binomial('logit'),data = 資料表),但如果自變數較多的話,前面那個運算式就會很長很長,萬一這些自變數還是基因名或編號,就很想死了。所以順便講一個簡化的辦法,即把那一串先寫成formula。
fml.std <- as.formula(paste('event~',
paste(colnames(egData)[c(5,11,13)],
collapse = '+')))
這裡有好幾層函式,paste() 會把括號中的元素貼上起來,collapse是其中的間隔。colnames() 是獲取表格的列名,[]中的數值向量為所選擇的列序號。這樣如果是一個超大表格,你選中第10~70列還可以寫成“10:70”。
好了,同樣寫出新模型的formula:
fml.new <- as.formula(paste('event~',
paste(colnames(egData)[c(5,11,13,19)],
collapse = '+')))
可以檢視一下,新模型的formula寫成這個效果:
然後像上邊說的那樣用glm()擬合兩個模型。
mstd = glm(fml.std, family = binomial('logit'),
data = egData, x=TRUE)
mnew = glm(fml.new, family = binomial('logit'),
data = egData, x=TRUE)
這樣一長串運算式用剛才命名好的fml.std和fml.new代替就好了。x=TRUE是將來用nricens包計算時要求用到的,表示輸出結果中是否包含所用到的資料表,平時可以不寫。
模型就這樣做完了~ 先不急著計算NRI,先看看它的總體情況。
summary(mstd)
執行這句就得到該模型的描述特徵。

殘差、相關係數、各個自變數的統計顯著性等,注意倒數第二行的AIC,就是上一期提到的赤池資訊準則,表示模型校準度,很少有人彙報呢。
可以用同樣的方法看看新模型。這裡就-不展開了,進入下一環節。
NRI的計算
• 先看nricens包的方法。
library(nricens)
NRI <- nribin(mdl.std = mstd, mdl.new = mnew,
updown = 'category',cut = c(0.3,0.6),
niter = 10000,alpha = 0.05)
填上新舊兩個模型。Cut是判斷風險高低的臨界值,現在我們寫了2個,也就是0~29%為低風險,30%~59%為中風險,60%~100%為高風險。現實中可以查閱相關文獻進行設定,預測風險達到多少需要怎樣干預之類的。
Updown為定義一個樣本的風險是否變動的方式,category是指分類值,即我就熟悉的低、中、高風險,另有一種diff,為連續值。選diff時,cut就設1個值,比如0.02,即認為當預測的風險在新舊模型中相差2%時,即被認為是重新分類了。這種方法用的比較少。
後面幾個引數就比較有意思了,niter為重複取樣的次數,即boostrap方法,不做的話將其設為0就好了;做的話建議至少1000次,這也是預設值,但我(讀書少)見過的研究都10000次。然後將統計顯著性alpha設為0.05。
這樣就可以看到輸出的結果:
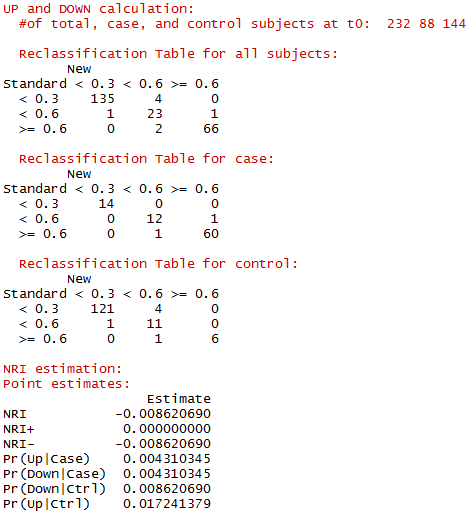
如果不做bootstrap,就是這個結果。有重新分類情況的詳表,最後是NRI和各種變動的概率。第一個NRI如前所述,是絕對NRI,大家可以根據之前的知識和上邊的詳表自己計算驗證一下,此時可手動計算出相對NRI。其他指標隨便看看。
如果做了bootstrap,就會多出一個表:

因為做了10000次重複取樣,相當於有10000個NRI,於是就有了標準誤和置信區間,剛才我們設alpha = 0.05,所以後面的Lower和Upper就是95%置信區間的下界和上界。
同時,做不做bootstrap都會得到一張圖,表示各資料點在新舊模型中的分佈。

預設的Case和Control標籤我覺得不太嚴謹,Case代表結局事件中編號為“1”的組,也就是發生了結局(死亡),Control為“0”,未發生。其實是positive和negative比較貼切吧。反正就這個意思。這張圖也和重新分類表的意思差不多,看看就好。
• 再看PredictABEL包的做法
library(PredictABEL)
pstd <- mstd$fitted.values
pnew <- mnew$fitted.values
先把兩個模型中的預測風險值提出來,也就是模型中的fitted.value。這個包只能從預測風險計算,剛才的nricens包可以用模型,也可以用預測風險(把mdl.std和mdl.new引數換成p.std和p.new)。
reclassification(data = egData,cOutcome = 21,
predrisk1 = pstd,predrisk2 = pnew,
cutoff = c(0,0.30,0.60,1))
cOutcome是結局事件的列序號,剛才我們不是把event放到最後了麼,即第21列,填上。兩個預測風險值也相應填上。這裡的cutoff跟剛才的不一樣,還要填上前面的0和後面的1,成為完整的0~100%的區間。
然後得到一個重新分類表:

跟上邊nricens做的差不多了。不過這個包沒有bootstrap的選項。
接著看下面的結果,這裡的分類NRI是咱們上回說的相加NRI,同樣可以根據上一期的知識手動計算一下。記得咱們並沒有設定bootstrap吧?可這裡也有個95%置信區間,只是內部呼叫了一個更為簡陋的只能計算連續NRI的improveProb()函式做的,而且連續變化的臨界值也不太透明,遂不管了。
最後還有個IDI是指,發生和未發生結局事件樣本的平均預測風險差異,在新模型中提高了0.44%。
延伸、總結和比較
這兩個包當然各有優劣。nricens計算時可控制的引數較多,彙報起來顯得華麗一些。PredictABEL計算結果則多了個IDI和大家喜聞樂見的p值。但也有學者表示,兩個模型的差異未必要求p<0.05。
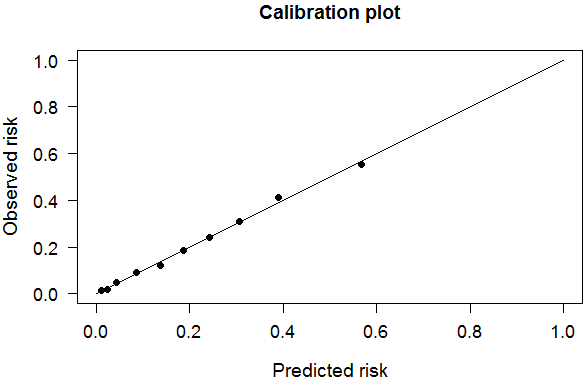
PredictABEL包還有很多有用的功能,比如可以做Hosmer- Lemeshow校準曲線,當然也附送p值(此處沒貼出來):
區分度箱形圖:

兩個模型的ROC曲線:
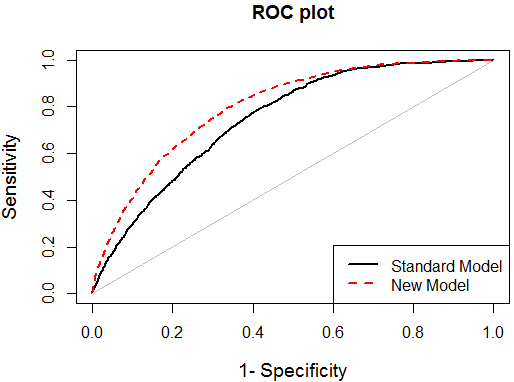
這都是評價一個模型很有價值的參考。
