python︱sklearn一些小技巧的記錄
sklearn裡面包含內容太多,所以一些實用小技巧還是挺好用的。
1、LabelEncoder
簡單來說 LabelEncoder 是對不連續的數字或者文字進行編號
from sklearn.preprocessing import LabelEncoder
le = LabelEncoder()
le.fit([1,5,67,100])
le.transform([1,1,100,67,5])
輸出: array([0,0,3,2,1])
2、OneHotEncoder
OneHotEncoder 用於將表示分類的資料擴維:
from sklearn.preprocessing import OneHotEncoder ohe = OneHotEncoder() ohe.fit([[1],[2],[3],[4]]) ohe.transform([2],[3],[1],[4]).toarray()
輸出:[ [0,1,0,0] , [0,0,1,0] , [1,0,0,0] ,[0,0,0,1] ]
正如keras中的keras.utils.to_categorical(y_train, num_classes)
.
3、sklearn.model_selection.train_test_split隨機劃分訓練集和測試集
一般形式:
train_test_split是交叉驗證中常用的函式,功能是從樣本中隨機的按比例選取train data和testdata,形式為:
X_train,X_test, y_train, y_test = cross_validation.train_test_split(train_data,train_target,test_size=0.4, random_state=0)
引數解釋:
- train_data:所要劃分的樣本特徵集
- train_target:所要劃分的樣本結果
- test_size:樣本佔比,如果是整數的話就是樣本的數量
- random_state:是隨機數的種子。
- 隨機數種子:其實就是該組隨機數的編號,在需要重複試驗的時候,保證得到一組一樣的隨機數。比如你每次都填1,其他引數一樣的情況下你得到的隨機陣列是一樣的。但填0或不填,每次都會不一樣。
隨機數的產生取決於種子,隨機數和種子之間的關係遵從以下兩個規則: - 種子不同,產生不同的隨機數;種子相同,即使例項不同也產生相同的隨機數。
fromsklearn.cross_validation import train_test_split train= loan_data.iloc[0: 55596, :] test= loan_data.iloc[55596:, :] # 避免過擬合,採用交叉驗證,驗證集佔訓練集20%,固定隨機種子(random_state) train_X,test_X, train_y, test_y = train_test_split(train, target, test_size = 0.2, random_state = 0) train_y= train_y['label'] test_y= test_y['label']
.
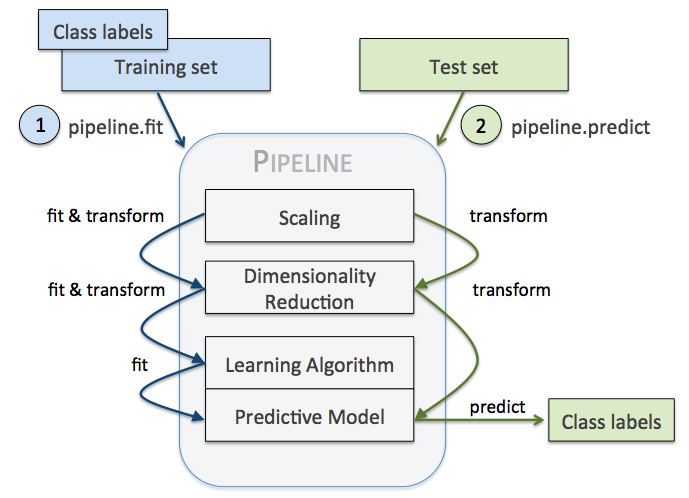
4、pipeline
本節參考與文章:用 Pipeline 將訓練集引數重複應用到測試集
pipeline 實現了對全部步驟的流式化封裝和管理,可以很方便地使引數集在新資料集上被重複使用。
pipeline 可以用於下面幾處:
- 模組化 Feature Transform,只需寫很少的程式碼就能將新的 Feature 更新到訓練集中。
- 自動化 Grid Search,只要預先設定好使用的 Model 和引數的候選,就能自動搜尋並記錄最佳的 Model。
- 自動化 Ensemble Generation,每隔一段時間將現有最好的 K 個 Model 拿來做 Ensemble。
問題是要對資料集 Breast Cancer Wisconsin 進行分類,
它包含 569 個樣本,第一列 ID,第二列類別(M=惡性腫瘤,B=良性腫瘤),
第 3-32 列是實數值的特徵。
from pandas as pd
from sklearn.cross_validation import train_test_split
from sklearn.preprocessing import LabelEncoder
df = pd.read_csv('https://archive.ics.uci.edu/ml/machine-learning-databases/'
'breast-cancer-wisconsin/wdbc.data', header=None)
# Breast Cancer Wisconsin dataset
X, y = df.values[:, 2:], df.values[:, 1]
encoder = LabelEncoder()
y = encoder.fit_transform(y)
>>> encoder.transform(['M', 'B'])
array([1, 0])
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=.2, random_state=0)
我們要用 Pipeline 對訓練集和測試集進行如下操作:
- 先用 StandardScaler 對資料集每一列做標準化處理,(是 transformer)
- 再用 PCA 將原始的 30 維度特徵壓縮的 2 維度,(是 transformer)
- 最後再用模型 LogisticRegression。(是 Estimator)
- 呼叫 Pipeline 時,輸入由元組構成的列表,每個元組第一個值為變數名,元組第二個元素是 sklearn 中的 transformer
或 Estimator。
注意中間每一步是 transformer,即它們必須包含 fit 和 transform 方法,或者 fit_transform。
最後一步是一個 Estimator,即最後一步模型要有 fit 方法,可以沒有 transform 方法。
然後用 Pipeline.fit對訓練集進行訓練,pipe_lr.fit(X_train, y_train)
再直接用 Pipeline.score 對測試集進行預測並評分 pipe_lr.score(X_test, y_test)
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
from sklearn.linear_model import LogisticRegression
from sklearn.pipeline import Pipeline
pipe_lr = Pipeline([('sc', StandardScaler()),
('pca', PCA(n_components=2)),
('clf', LogisticRegression(random_state=1))
])
pipe_lr.fit(X_train, y_train)
print('Test accuracy: %.3f' % pipe_lr.score(X_test, y_test))
# Test accuracy: 0.947
還可以用來選擇特徵:
例如用 SelectKBest 選擇特徵,
分類器為 SVM,
anova_filter = SelectKBest(f_regression, k=5)
clf = svm.SVC(kernel='linear')
anova_svm = Pipeline([('anova', anova_filter), ('svc', clf)])
當然也可以應用 K-fold cross validation:
model = Pipeline(estimators)
seed = 7
kfold = KFold(n_splits=10, random_state=seed)
results = cross_val_score(model, X, Y, cv=kfold)
print(results.mean())
Pipeline 的工作方式
當管道 Pipeline 執行 fit 方法時,
首先 StandardScaler 執行 fit 和 transform 方法,
然後將轉換後的資料輸入給 PCA,
PCA 同樣執行 fit 和 transform 方法,
再將資料輸入給 LogisticRegression,進行訓練。
5 稀疏矩陣合併
from scipy.sparse import hstack
x_train_spar = hstack([x_train_ens_s, x_train_tfidf])
hstack([X, csr_matrix(char_embed), csr_matrix(word_embed)], format='csr')
多項式PolynomialFeatures,有點類似FFM模型,可以組合特徵
poly = PolynomialFeatures(degree=2, interaction_only=True, include_bias=False)
#degree控制多項式最高次數
x_train_new = poly.fit_transform(x_train)