使用Python對大腦成像資料進行視覺化分析
## 簡介
大腦是人類目前所知的最複雜的器官,為了很好的瞭解大腦這個器官,我們做了很多努力,核磁共振成像(Magnetic Resonance Image,MRI)技術就是其中的重要突破,通過MRI的方式,我們可以獲得大腦的一些資料。
近年來,隨著機器學習的興起,醫學資料與機器學習結合使用的情況越來越多,而要有效的使用好醫學資料,其前提就是處理好這些資料,本文內容會重點介紹如何使用Python來處理與分析這些腦成像資料,不會涉及過多醫學知識。
## sMRI與fMRI
腦成像相關的資料可以去SPM網站中下載,SPM的含義是統計引數對映(Statistical Paramtric Mapping),MRI生成的資料其實就是一種引數對映資料,當然,更加方便的是在工作公眾號中回覆data3獲得相應的資料與jupyter程式碼檔案。
下載後,其中有4個檔案,其中README開頭的為描述檔案,fM00223為功能性核磁共振(funciton MRI,fMRI)成像資料,sM00223為結構性核磁共振(structural MRI, sMRI)成像資料。通過描述檔案可知,這些資料是一個人躺在MRI機器上聽「雙音節詞」時大腦的成像資料。
為了方便理解後面資料處理的內容,有必要理解sMRI與fMRI是什麼以及兩者的差異。
### 結構性核磁共振(sMRI)
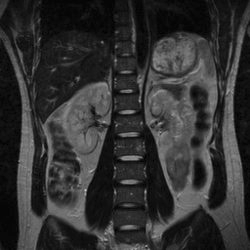
因為人的體記憶體有大量的水分子,而水分子中還有氫原子,sMRI其實就是利用氫原子來成像,這意味著人身體中的內臟、軟組織等含有高水分與脂肪的器官會被清楚的掃描出來,而大腦就是這樣的一個器官,通過sMRI可以清晰的看到大腦中的密集結構與大量細節,但sMRI的成像無法觀察到大腦的運動情況,即無法判斷那些部位目前是比較活躍的,只能給出大腦的結構細節。
如下圖,科學家利用sMRI對人體腹腔進行成像,從圖可以看出,腹腔的結構很明顯。
### 功能性核磁共振(fMRI)
為了彌補sMRI的缺陷,fMRI應運而出,fMRI主要通過血氧濃度水平依賴(Blood-oxygen-level dependent,BOLD)來成像,一個器官的某個部位活躍,此時這個器官的這個部位就需要消耗更多的氧氣,以此為依據,來進行成像。
通過fMRI的方式,我們可以很好的判斷出大腦此時那些區域是活躍的,但這種活動並不等同於神經活動,但fMRI也有缺陷,即它的成像會損失大量的細節。
如下圖,科學家通過fMRI,利用BOLD探索大腦活躍區域。
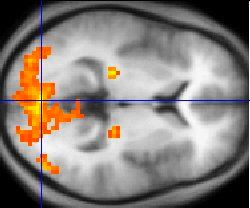
但從圖中可以看出,大腦的細節幾乎看不清晰,所以目前常規的方式是使用sMRI對大腦結構進行成像,而fMRI對大腦活躍區域進行成像。
## sMRI資料視覺化處理
通常,神經影像檔案都會以相應的規律將資料儲存在固定檔案格式的檔案中,我們可以通過NiBabel庫來讀/寫常見的神經影像檔案中的資料。
sMRI對應的資料在sM00223資料夾下,進入資料夾可以發現有兩種不同檔案格式的資料,分別是.img與.hdr,這其實是醫學影像領域常見的格式,主要用於「分析」,其中,.img作為資料檔案,包含二進位制的影象資料,而.hdr作為標頭檔案,包含影象的元資料,但這兩種格式現在逐漸被.nifti格式代替,這是因為.hdr標頭檔案難以完全真實反映元資料。
使用NiBabel將sMRI獲得的資料載入:
import nibabel
\# sM0223對應的檔案
data\_path = './fMRI\_data/sM00223/'
files = os.listdir(data_path)
\# 讀取其中的資料
data_all = \[\]
for data_file in files:
if data_file\[-3:\] == 'hdr':
data = nibabel.load(data\_path + data\_file).get_data()
\# 列印資料維數
print(data.shape)
\# -------結果
(256, 256, 54, 1)
可以看出,sMRI產生的是4維資料,但第4維其實沒有包含任何資訊,其說明了sMRI每次掃描會產生54個數據切片,每個切片對應影象的大小為256x256個體素。
> 體素:類似畫素,畫素表示的是二維影象的最小單位,而體素則用於三維成像空間。
為了方面視覺化每個切片的資料,可以簡單處理一下資料:
import numpy as np
data = np.rot90(data.squeeze(), 1)
print(data.shape)
\# -------結果
(256, 256, 54)
上述程式碼中,先通過numpy.squeeze()刪除了陣列中的單維條目,此時無用的第四維會被刪除,接著使用numpy.rot90()將陣列逆時針旋轉了90度。
簡單處理後,直接使用Matplotlib對每10個切片中的一個切片進行資料的繪製。
import matplotlib.pyplot as plt
\# 建立 6 個子圖,不清楚其中概念,可以看本公眾號關於Matplotlib的文章
fig, ax = plt.subplots(1, 6, figsize=\[18, 3\])
n = 0
slice = 0
for _ in range(6):
ax\[n\].imshow(data\[:, :, slice\], 'gray')
ax\[n\].set_xticks(\[\])
ax\[n\].set_yticks(\[\])
ax\[n\].set_title('Slice number: {}'.format(slice), color='r')
n += 1
slice += 10
fig.subplots_adjust(wspace=0, hspace=0)
plt.show()
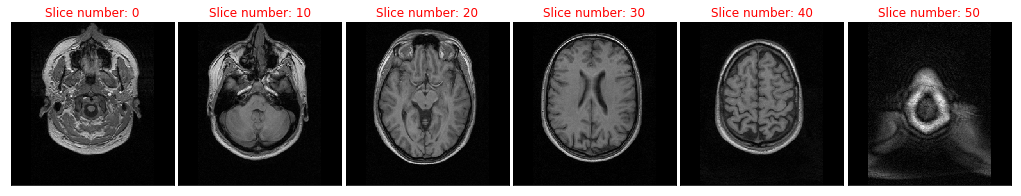
這就是通過sMRI資料繪製出的大腦結構圖了,其中第0層切片是最低的一個(接近脖子位置),而第50層切片是最高的一個(接近頭頂),在第20層,可以看具有眼睛的切片。
## fMRI資料視覺化處理
閱讀README.txt可知fM00223資料集中,每張影象的大小為64x64個體素,採集的片數為64以及採集的卷數為96。知道了這些資訊,就可以對fM00223資料集中的資料進行重構。
開啟fM00223資料夾,可以發現確實正好有96個Hdr檔案,這意味著每個檔案包含了一個卷的所有片。
\# fMRI資料的基本資訊
x_size = 64
y_size = 64
n_slice = 64
n_volumes = 96
\# 獲得所有檔案
data\_path = './fMRI\_data/fM00223/'
files = os.listdir(data_path)
\# 讀取資料並進行重塑
data_all = \[\]
for data_file in files:
if data_file\[-3:\] == 'hdr':
data = nibabel.load(data\_path + data\_file).get_data()
# 將所有資料新增到list中,從而多了一個維度:時間維度
data\_all.append(data.reshape(x\_size, y\_size, n\_slice))
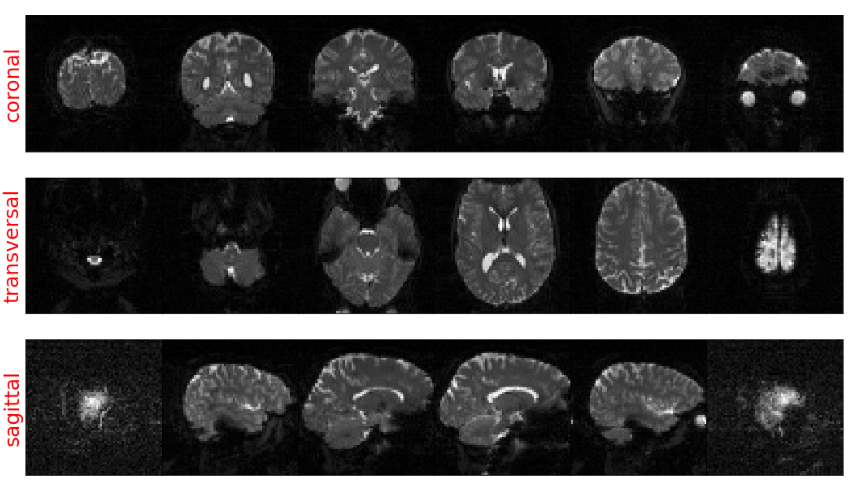
接著就可以通過Matplotlib視覺化展示資料了,因為組成這些資料的是體素,是三維的影象,我們無法直接看到所有的資料,所有進行切片操作,通常會橫切大腦從而獲得冠狀面(coronal)、橫切面(transversal)和矢狀面(sagittal)這3個平面,理解這三個概念很重要,如下圖:
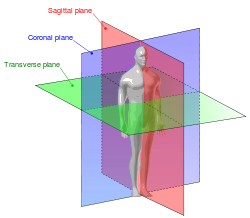
+ 冠狀面為左,右方向將人體縱切為前後兩部分的斷面
+ 橫切面指橫向水平切割的平面
+ 矢狀面是指將軀體縱斷為左右兩部分的解剖平面
看一下下面的程式碼:
\# 建立 3x6 的子圖,大小為 18x11
fig, ax = plt.subplots(3, 6, figsize=\[18, 11\])
\# 組織資料以冠狀平面進行視覺化,第四維度為時間維度,這裡取第一個時間點
coronal = np.transpose(data_all, \[1, 3, 2, 0\])
coronal = np.rot90(coronal, 1)
\# 組織資料以橫切平面進行視覺化
transversal = np.transpose(data_all, \[2, 1, 3, 0\])
transversal = np.rot90(transversal, 2)
\# 組織資料以矢狀平面進行視覺化
sagittal = np.transpose(data_all, \[2, 3, 1, 0\])
sagittal = np.rot90(sagittal, 1)
\# 視覺化不同平面
n = 10
\# 對每個切片的6個切面進行操作
for i in range(6):
ax\[0\]\[i\].imshow(coronal\[:, :, n, 0\], cmap='gray')
ax\[0\]\[i\].set_xticks(\[\])
ax\[0\]\[i\].set_yticks(\[\])
if i == 0:
ax\[0\]\[i\].set_ylabel('coronal', fontsize=25, color='r')
n += 10
n = 5
for i in range(6):
ax\[1\]\[i\].imshow(transversal\[:, :, n, 0\], cmap='gray')
ax\[1\]\[i\].set_xticks(\[\])
ax\[1\]\[i\].set_yticks(\[\])
if i == 0:
ax\[1\]\[i\].set_ylabel('transversal', fontsize=25, color='r')
n += 10
n = 5
for i in range(6):
ax\[2\]\[i\].imshow(sagittal\[:, :, n, 0\], cmap='gray')
ax\[2\]\[i\].set_xticks(\[\])
ax\[2\]\[i\].set_yticks(\[\])
if i == 0:
ax\[2\]\[i\].set_ylabel('sagittal', fontsize=25, color='r')
n += 10
fig.subplots_adjust(wspace=0, hspace=0)
plt.show()
## 尾
感謝閱讀,如果喜歡,點好看關注,如果有很想跟我說的話,歡迎直接跟我對話,個人微信:sighblue。
