TPM、read counts、RPKM/FPKM你選對了嗎?
TPM、read counts、RPKM/FPKM你選對了嗎?
本文轉載自嘉因微信公眾號,已獲得授權。檢視最新文章,敬請關注嘉因,微信ID:rainbow-genome
作者:小哈 來源:嘉因
在RNA測序分析,哪家公司適合我一文中,聊過類似的問題。
這次小哈搬來了statQuest的3個最新視訊,https://statquest.org/video-index/,點選左下角“閱讀原文”直達statQuest視訊目錄。幫你理清RNA-seq資料預處理方法,哪個更適合你的問題。
先說結論:
-
學術界已經不再推薦RPKM、FPKM;
-
比較基因的表達丰度,例如哪個基因在哪個組織裡高表達,用TPM做均一化處理;
-
不同組間比較,找差異基因,先得到read counts,然後用DESeq2或edgeR,做均一化和差異基因篩選;如果對比某個基因的KO組和對照,推薦DESeq2。
如果找公司做RNA-seq資料處理,計算表達量時,記得要read counts。
RPKM/FPKM、TPM
在RNA-Seq分析|RPKM, FPKM, TPM, 傻傻分不清楚?
下面是視訊截圖
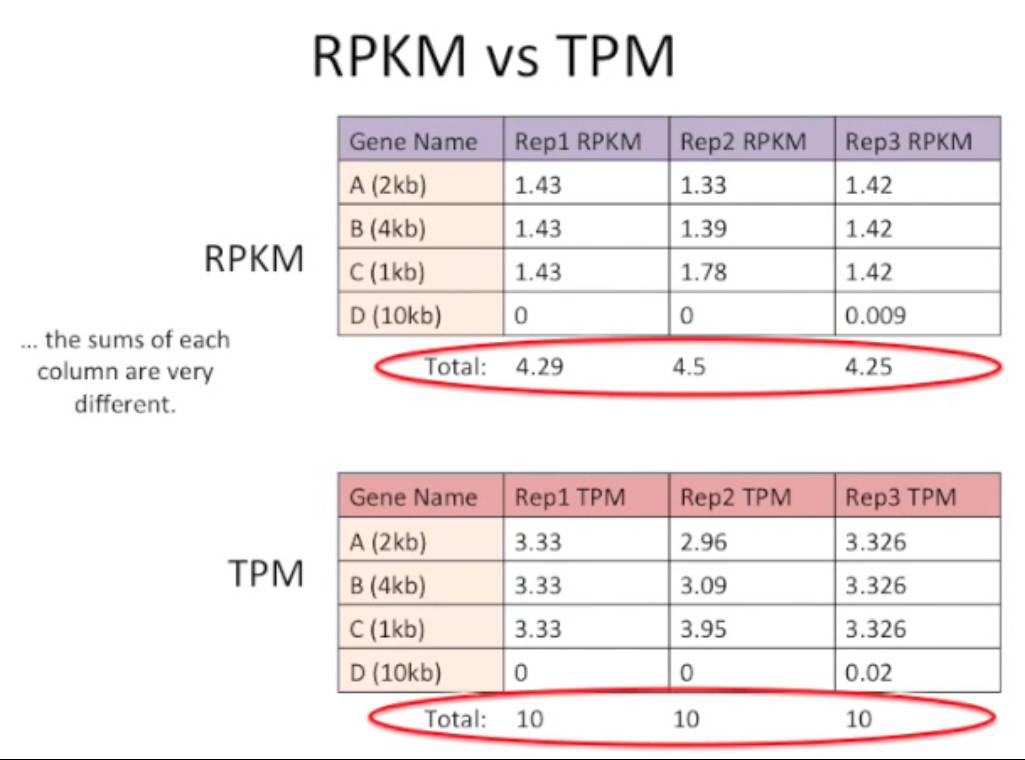
不再用RPKM/FPKM,現在推薦用TPM

一表看懂TPM更適合比較同一基因在不同sample間表達丰度的差異
DESeq2或edgeR
DESeq2和edgeR不用RPKM/FPKM或TPM做均一化,而是直接用原始的read counts做均一化處理。

DESeq2和edgeR能很好的解決這兩個問題

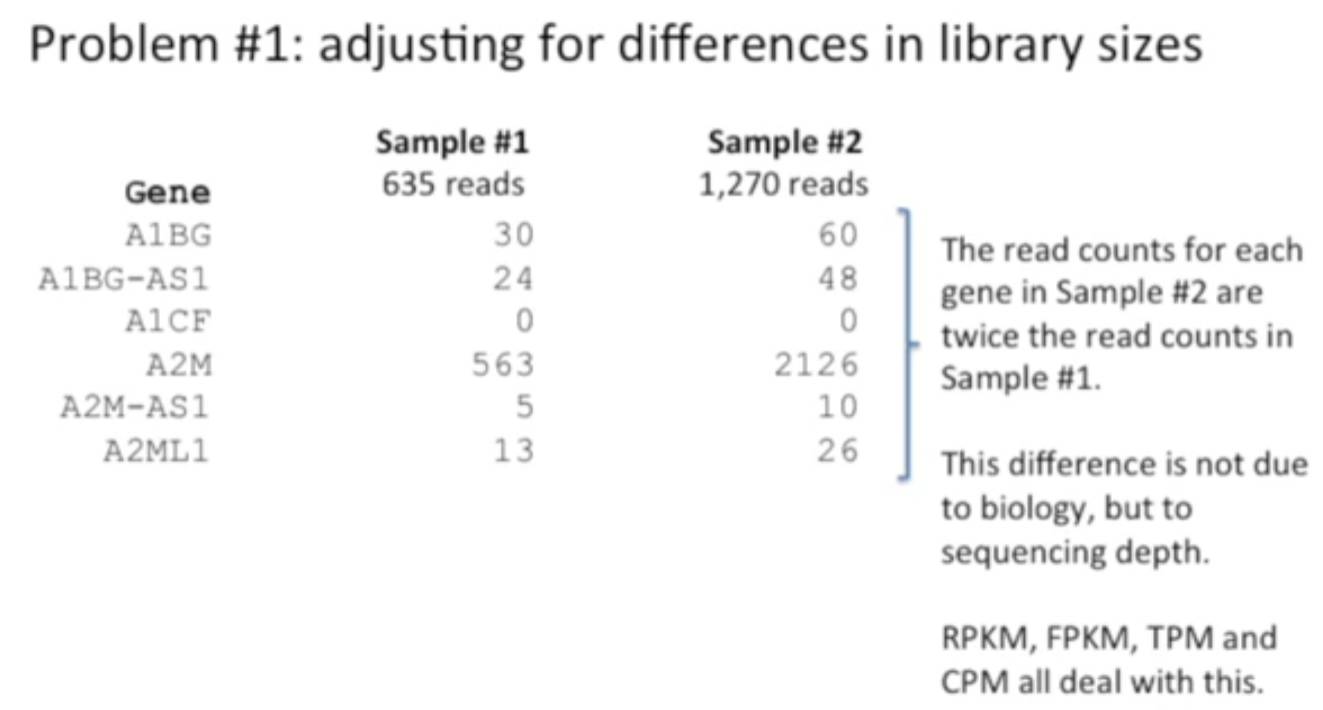
測序深度的差異問題,用RPKM/FPKM、TPM、DESeq2和edgeR都能處理
如果組間RNA成分差異較大,怎麼辦?
例如liver跟spleen比,組織特異性表達基因在裡面搗亂;再例如常見的某個基因KO組與對照相比,該基因成分在KO組裡缺失。



DESeq2做均一化
用edgeR作均一化
