資料探勘工具---spark使用練習---ml(一)
Spark中ml和mllib的區別
來源:
Spark中ml和mllib的主要區別和聯絡如下:
- ml和mllib都是Spark中的機器學習庫,目前常用的機器學習功能2個庫都能滿足需求。
- spark官方推薦使用ml, 因為ml功能更全面更靈活,未來會主要支援ml,mllib很有可能會被廢棄(據說可能是在spark3.0中deprecated)。
- ml主要操作的是DataFrame, 而mllib操作的是RDD,也就是說二者面向的資料集不一樣。相比於mllib在RDD提供的基礎操作,ml在DataFrame上的抽象級別更高,資料和操作耦合度更低。
- DataFrame和RDD什麼關係?DataFrame是Dataset的子集,也就是Dataset[Row], 而DataSet是對RDD的封裝,對SQL之類的操作做了很多優化。
- 相比於mllib在RDD提供的基礎操作,ml在DataFrame上的抽象級別更高,資料和操作耦合度更低。
- ml中的操作可以使用pipeline, 跟sklearn一樣,可以把很多操作(演算法/特徵提取/特徵轉換)以管道的形式串起來,然後讓資料在這個管道中流動。大家可以腦補一下Linux管道在做任務組合時有多麼方便。
- ml中無論是什麼模型,都提供了統一的演算法操作介面,比如模型訓練都是fit;不像mllib中不同模型會有各種各樣的trainXXX。
- mllib在spark2.0之後進入維護狀態, 這個狀態通常只修復BUG不增加新功能。
以上就是ml和mllib的主要異同點。下面是ml和mllib邏輯迴歸的例子,可以對比看一下, 雖然都是模型訓練和預測,但是畫風很不一樣。
Spark ML中的機器學習
PySpark.ML模組介紹
ML模組包括機器學習三個核心功能:
(1)資料準備: 特徵提取、變換、選擇、分類特徵的雜湊和自然語言處理等等;
(2)機器學習方法: 實現了一些流行和高階的迴歸,分類和聚類演算法;
(3)實用程式: 統計方法,如描述性統計、卡方檢驗、線性代數(稀疏稠密矩陣和向量)和模型評估方法。
目前ML模組還處於不斷髮展中,但是已經可以滿足我們的基礎資料科學任務。
注意:官網上,標註“E”為測試階段,不穩定,可能會產生錯誤失敗。
ML模組三個抽象類:
轉換器(Transformer)、評估器(Estimator)和管道(Pipeline)
資料準備
資料檢視
df.describe().show()
資料的檢視和轉換操作很多都可以利用pyspark.sql下的api介面來實現,比如一般資料預處理時候,我們會用pyspark.ml.feature module中的介面來實現,但如果不能滿足,其實pyspark.sql.functions介面可能實現更多的變換。我們只要先對某一列變換,然後再加回到df.就好比在sklearn中我們會優先使用sklearn.preprocessing來完成預處理,但如果不能夠滿足自己的要求,我個人會使用map函式來做變換。
資料型別的轉換
首先是在讀入資料時,可以根據實際情形,設定inferschema=True,如
df = spark.read.csv(path='/tmp/test/hour.csv', header=True, sep=',',inferSchema=True)
這樣會自動判斷型別,否則會部為string型別。
但是自動識別型別,有時還是不能滿足需求,就需要人為轉換,目前還沒找到方法,不過應該是可以的。
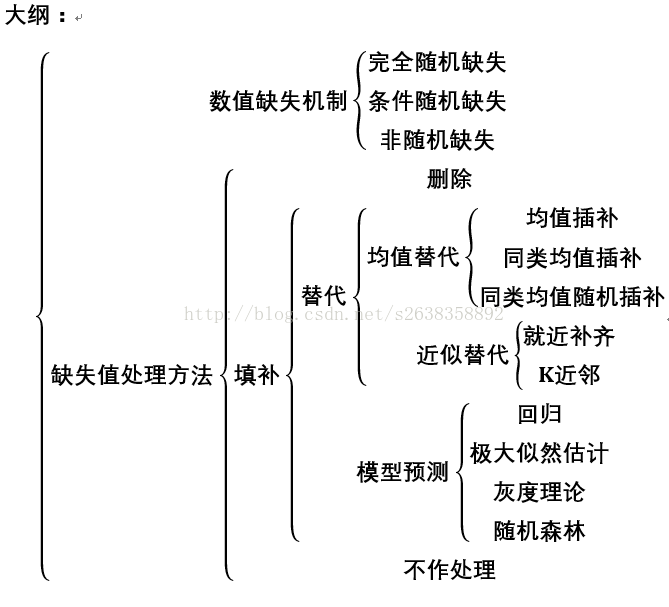
缺失值處理
pyspark中檢視缺失值似乎並沒有太好的方法,但可以反覆用df.desribe().show()或df.summary().show()來檢視,因為對於’’,null是當成字元來看,與數字混在一起,要麼最小,最麼最最大,然後統計的時候是被排除在外的;NaN被當成最大,統計出來的均值、方差也是NaN。如下
df=spark.sql('select * from pankoo.test')
df.show()
"""
+-----+-----+-----+
|name1|name2|name3|
+-----+-----+-----+
| 1| | 2|
| 3| 4| NaN|
| Null| 5| 6|
+-----+-----+-----+
"""
df.describe().show()
"""
+-------+------------------+------------------+-----+
|summary| name1| name2|name3|
+-------+------------------+------------------+-----+
| count| 3| 3| 3|
| mean| 2.0| 4.5| NaN|
| stddev|1.4142135623730951|0.7071067811865476| NaN|
| min| 1| | 2|
| max| Null| 5| NaN|
+-------+------------------+------------------+-----+
"""
對於離散型變數,可以用select distinct xx from 。
如果是標準的用nan表示缺失值,也可像下面那樣用df.filter來檢視。或者不用檢視,直接進行填充。
for i in range(len(fieldIndex)):
df.filter('%s is null'%(fieldIndex[i])).select(fieldIndex[i]).limit(10).show()
整理來講在分散式環境下檢視資料代價還是挺大的。
利用pyspark.sql.DataFrame的介面
利用類似下面的語句進行缺少值填充
df.na.fill({'age': 50, 'name': 'unknown'})
具體填充的值,可用類似下面的語句獲取。
#mean方法只接受數值型別的引數,Int, Long等,如果是String, Date, Timestamp 型別的話要用agg(mean(“b”))
meanTemp=df.agg({'temp': 'avg'}).collect()[0][0]
print(meanTemp)
for i in range(len(fieldIndex)):
meanTemp = df.agg({fieldIndex[i]: 'avg'}).collect()[0][0]
df.na.fill({fieldIndex[i]:meanTemp})
也可以用pyspark.ml.feature.Imputer(*args, **kwargs)來實現缺失值的填充
類似於
imputer=Imputer(strategy='mean',inputCols=['atemp'],outputCols=['out_atemp'])
model=imputer.fit(df)
df=model.transform(df)
轉換器
pyspark.ml.Transformer
通常通過將一個新列附加到DataFrame來轉換資料。
當從轉換器的抽象類派生時,每個新的轉換器類需要實現.transform()方法,該方法要求傳遞一個要被轉換的DataFrame,該引數通常是第一個也是唯一的一個強制性引數。
transform(dataset, params=None)
Transforms the input dataset with optional parameters(使用可選引數轉換輸入資料集。).
Parameters:
**dataset** – input dataset, which is an instance of pyspark.sql.DataFrame
**params** – an optional param map that overrides embedded params.
Returns: transformed dataset
pyspark.ml.feature模組提供了許多的轉換器:
pyspark.ml.feature.Binarizer(self, threshold=0.0, inputCol=None, outputCol=None)
根據指定的閾值將連續變數轉換為對應的二進位制
#-*-coding:utf-8-*-
from pyspark import SparkContext
from pyspark.sql import SparkSession
from pyspark.ml.feature import Binarizer
if __name__=="__main__":
sc=SparkContext(appName="myApp")
spark=SparkSession.builder.enableHiveSupport().getOrCreate()
df=spark.createDataFrame([(0.5,)],["values"])#注意0.5後面有個逗號,不加會出錯,不知道為什麼要這樣;
"""
df.show()
+------+
| values |
+------+
| 0.5 |
+------+
"""
binarizer=Binarizer(threshold=1.0,inputCol="values",outputCol="features")
bd=binarizer.transform(df)
"""
bd.show()
+------+-------+
| values | features |
+------+-------+
| 0.5 | 0.0 |
+------+-------+
print(bd.head().features)
0.0
"""
binarizer.setParams(outputCol="freqs")
bdf=binarizer.transform(df)
"""
bdf.show()
+------+-----+
| values | freqs |
+------+-----+
| 0.5 | 0.0 |
+------+-----+
"""
params={binarizer.threshold:-0.5,binarizer.outputCol:"vectors"}
bdf=binarizer.transform(df,params=params)
"""
bdf.show()
+------+-------+
| values | vectors |
+------+-------+
| 0.5 | 1.0 |
+------+-------+
"""
temp_path="/tmp/test/"
binarizerPath ="%sbinarizer"%(temp_path)
binarizer.save(binarizerPath)
loadBinarizer=Binarizer.load(binarizerPath)
# print(loadBinarizer.getThreshold()==binarizer.getThreshold())
# True
pyspark.ml.feature.Bucketizer(self, splits=None, inputCol=None, outputCol=None, handleInvalid="error")
與Binarizer類似,該方法根據閾值列表(分割的引數),將連續變數轉換為多項值(連續變數離散化到指定的範圍區間)
#-*-coding:utf-8-*-
from pyspark import SparkContext
from pyspark.sql import SparkSession
from pyspark.ml.feature import Bucketizer
import numpy as np
if __name__=="__main__":
sc=SparkContext(appName="myApp")
spark=SparkSession.builder.getOrCreate()
values=[(0.1,),(0.4,),(1.2,),(1.5,),(np.nan,),(np.nan,)]
# values = [(0.1,), (0.4,), (1.2,), (1.5,), (float("nan"),), (float("nan"),)]
df=spark.createDataFrame(values,["values"])
"""
df.show()
+------+
|values|
+------+
| 0.1|
| 0.4|
| 1.2|
| 1.5|
| NaN|
| NaN|
+------+
"""
bucketizer=Bucketizer(splits=[-float("inf"),0.5,1.4,float("inf")],inputCol="values",outputCol="buckets",handleInvalid="keep")
bdf=bucketizer.transform(df)
"""
bdf.show()
+------+-------+
| values | buckets |
+------+-------+
| 0.1 | 0.0 |
| 0.4 | 0.0 |
| 1.2 | 1.0 |
| 1.5 | 2.0 |
| NaN | 3.0 |
| NaN | 3.0 |
print(bdf.head().buckets)
0.0
print(bdf.collect()[0].buckets)
0.0
print(bdf.collect()[3].buckets)
2.0
"""
bucketizer.setParams(outputCol="b")
bdf=bucketizer.transform(df)
"""
bdf.show()
+------+---+
| values | b |
+------+---+
| 0.1 | 0.0 |
| 0.4 | 0.0 |
| 1.2 | 1.0 |
| 1.5 | 2.0 |
| NaN | 3.0 |
| NaN | 3.0 |
+------+---+
"""
temp_path = "/tmp/test/"
bucketizerPath ="%sbucketizer"%(temp_path)
bucketizer.save(bucketizerPath)
loadBucketizer=Bucketizer.load(bucketizerPath)
# print(loadBucketizer.getSplits()==bucketizer.getSplits() )
# True
pyspark.ml.feature.ChiSqSelector(self, numTopFeatures=50, featuresCol="features", outputCol=None, labelCol="label", selectorType="numTopFeatures", percentile=0.1, fpr=0.05, fdr=0.05, fwe=0.05)
對於分類目標變數(思考分類模型),此功能允許你選擇預定義數量的特徵(由numTopFeatures引數進行引數化),以便最好地說明目標的變化。該方法需要兩部:需要.fit()——可以計算卡方檢驗,呼叫.fit()方法,將DataFrame作為引數傳入返回一個ChiSqSelectorModel物件,然後可以使用該物件的.transform()方法來轉換DataFrame。預設情況下,選擇方法是numTopFeatures,預設頂級要素數設定為50。
if __name__=="__main__":
sc=SparkContext(appName="myApp")
spark=SparkSession.builder.getOrCreate()
df=spark.createDataFrame([(Vectors.dense([0.0, 0.0, 18.0, 1.0]),1.0),(Vectors.dense([0.0, 1.0, 12.0, 0.0]),0.0),(Vectors.dense([1.0, 0.0, 15.0, 0.1]),0.0)],["features","label"])
"""
df.show()
+------------------+-----+
| features | label |
+------------------+-----+
| [0.0, 0.0, 18.0, 1.0] | 1.0 |
| [0.0, 1.0, 12.0, 0.0] | 0.0 |
| [1.0, 0.0, 15.0, 0.1] | 0.0 |
+------------------+-----+
"""
chiSqSelector=ChiSqSelector(numTopFeatures=1,outputCol="selectedFeatures")
model=chiSqSelector.fit(df)
# print(model.selectedFeatures)
# [2]
cdf=model.transform(df)
"""
cdf.show()
+------------------+-----+----------------+
| features | label | selectedFeatures |
+------------------+-----+----------------+
| [0.0, 0.0, 18.0, 1.0] | 1.0 | [18.0] |
| [0.0, 1.0, 12.0, 0.0] | 0.0 | [12.0] |
| [1.0, 0.0, 15.0, 0.1] | 0.0 | [15.0] |
+------------------+-----+----------------+
"""
pyspark.ml.feature.CountVectorizer(self, minTF=1.0, minDF=1.0, vocabSize=1 << 18, binary=False, inputCol=None, outputCol=None)
用於標記文字
>>> df = spark.createDataFrame(
... [(0, ["a", "b", "c"]), (1, ["a", "b", "b", "c", "a"])],
... ["label", "raw"])
>>> cv = CountVectorizer(inputCol="raw", outputCol="vectors")
>>> model = cv.fit(df)
>>> model.transform(df).show(truncate=False)
+-----+---------------+-------------------------+
|label|raw |vectors |
+-----+---------------+-------------------------+
|0 |[a, b, c] |(3,[0,1,2],[1.0,1.0,1.0])|
|1 |[a, b, b, c, a]|(3,[0,1,2],[2.0,2.0,1.0])|
+-----+---------------+-------------------------+
...
>>> sorted(model.vocabulary) == ['a', 'b', 'c']
True
>>> countVectorizerPath = temp_path + "/count-vectorizer"
>>> cv.save(countVectorizerPath)
>>> loadedCv = CountVectorizer.load(countVectorizerPath)
>>> loadedCv.getMinDF() == cv.getMinDF()
True
>>> loadedCv.getMinTF() == cv.getMinTF()
True
>>> loadedCv.getVocabSize() == cv.getVocabSize()
True
>>> modelPath = temp_path + "/count-vectorizer-model"
>>> model.save(modelPath)
>>> loadedModel = CountVectorizerModel.load(modelPath)
>>> loadedModel.vocabulary == model.vocabulary
True
pyspark.ml.feature.Imputer(*args, **kwargs)
用於完成缺失值的插補估計器,使用缺失值所在列的平均值或中值。 輸入列應該是DoubleType或FloatType。 目前的Imputer不支援分類特徵,可能會為分類特徵建立不正確的值。
請注意,平均值/中值是在過濾出缺失值之後計算的。 輸入列中的所有Null值都被視為缺失,所以也被歸類。 為了計算中位數,使用pyspark.sql.DataFrame.approxQuantile(),相對誤差為0.001。
#-*-coding:utf-8-*-
from pyspark import SparkContext
from pyspark.sql import SparkSession
from pyspark.ml.feature import Imputer
from pyspark.ml.feature import ImputerModel
if __name__=="__main__":
sc=SparkContext(appName="myApp")
spark=SparkSession.builder.getOrCreate()
df=spark.createDataFrame([(1.0,float("nan")),(2.0,float("nan")),(float("nan"),3.0),(4.0,4.0),(5.0,5.0)],["a","b"])
"""
df.show()
+---+---+
| a | b |
+---+---+
| 1.0 | NaN |
| 2.0 | NaN |
| NaN | 3.0 |
| 4.0 | 4.0 |
| 5.0 | 5.0 |
+---+---+
"""
imputer=Imputer(inputCols=["a","b"],outputCols=["out_a","out_b"])
model=imputer.fit(df)
"""
model.surrogateDF.show()
#預設情況下,就是取出了ab兩列的均值
+---+---+
| a | b |
+---+---+
| 3.0 | 4.0 |
+---+---+
"""
idf=model.transform(df)
"""
idf.show()
+---+---+-----+-----+
| a | b | out_a | out_b |
+---+---+-----+-----+
| 1.0 | NaN | 1.0 | 4.0 |
| 2.0 | NaN | 2.0 | 4.0 |
| NaN | 3.0 | 3.0 | 3.0 |
| 4.0 | 4.0 | 4.0 | 4.0 |
| 5.0 | 5.0 | 5.0 | 5.0 |
+---+---+-----+-----+
"""
imputer.setStrategy("median").setMissingValue(1.0)
model=imputer.fit(df)
idf=model.transform(df)
"""
idf.show()
+---+---+-----+-----+
| a | b | out_a | out_b |
+---+---+-----+-----+
| 1.0 | NaN | 4.0 | NaN |
| 2.0 | NaN | 2.0 | NaN |
| NaN | 3.0 | NaN | 3.0 |
| 4.0 | 4.0 | 4.0 | 4.0 |
| 5.0 | 5.0 | 5.0 | 5.0 |
+---+---+-----+-----+
"""
temp_path="/tmp/test/"
imputer_path="%simputer"%(temp_path)
imputer.save(imputer_path)
loadImputer=Imputer.load(imputer_path)
# print(loadImputer.getStrategy()==imputer.getStrategy())
# True
model_path="%simputermodel"%(temp_path)
model.save(model_path)
loadModel=ImputerModel.load(model_path)
# print(loadModel.transform(df).head().out_a==model.transform(df).head().out_a)
# True
pyspark.ml.feature.MaxAbsScaler(self, inputCol=None, outputCol=None)
通過分割每個特徵中的最大絕對值來單獨重新縮放每個特徵以範圍[-1,1]。 它不會移動/居中資料,因此不會破壞任何稀疏性。
>>> from pyspark.ml.linalg import Vectors
>>> df = spark.createDataFrame([(Vectors.dense([1.0]),), (Vectors.dense([2.0]),)], ["a"])
>>> maScaler = MaxAbsScaler(inputCol="a", outputCol="scaled")
>>> model = maScaler.fit(df)
>>> model.transform(df).show()
+-----+------+
| a|scaled|
+-----+------+
|[1.0]| [0.5]|
|[2.0]| [1.0]|
+-----+------+
...
>>> scalerPath = temp_path + "/max-abs-scaler"
>>> maScaler.save(scalerPath)
>>> loadedMAScaler = MaxAbsScaler.load(scalerPath)
>>> loadedMAScaler.getInputCol() == maScaler.getInputCol()
True
>>> loadedMAScaler.getOutputCol() == maScaler.getOutputCol()
True
>>> modelPath = temp_path + "/max-abs-scaler-model"
>>> model.save(modelPath)
>>> loadedModel = MaxAbsScalerModel.load(modelPath)
>>> loadedModel.maxAbs == model.maxAbs
True
pyspark.ml.feature.MinMaxScaler(self, min=0.0, max=1.0, inputCol=None, outputCol=None)
使用列彙總統計資訊,將每個特徵單獨重新標定為一個常用範圍[min,max],這也稱為最小 - 最大標準化或重新標定(注意由於零值可能會被轉換為非零值,因此即使對於稀疏輸入,轉換器的輸出也將是DenseVector)。 特徵E的重新縮放的值被計算為,資料將被縮放到[0.0,1.0]範圍內。For the case E_max == E_min, Rescaled(e_i) = 0.5 * (max + min)```
>>> from pyspark.ml.linalg import Vectors
>>> df = spark.createDataFrame([(Vectors.dense([0.0]),), (Vectors.dense([2.0]),)], ["a"])
>>> mmScaler = MinMaxScaler(inputCol="a", outputCol="scaled")
>>> model = mmScaler.fit(df)
>>> model.originalMin
DenseVector([0.0])
>>> model.originalMax
DenseVector([2.0])
>>> model.transform(df).show()
+-----+------+
| a|scaled|
+-----+------+
|[0.0]| [0.0]|
|[2.0]| [1.0]|
+-----+------+
...
>>> minMaxScalerPath = temp_path + "/min-max-scaler"
>>> mmScaler.save(minMaxScalerPath)
>>> loadedMMScaler = MinMaxScaler.load(minMaxScalerPath)
>>> loadedMMScaler.getMin() == mmScaler.getMin()
True
>>> loadedMMScaler.getMax() == mmScaler.getMax()
True
>>> modelPath = temp_path + "/min-max-scaler-model"
>>> model.save(modelPath)
>>> loadedModel = MinMaxScalerModel.load(modelPath)
>>> loadedModel.originalMin == model.originalMin
True
>>> loadedModel.originalMax == model.originalMax
True
pyspark.ml.feature.Normalizer(self, p=2.0, inputCol=None, outputCol=None)
使用給定的p範數標準化向量以得到單位範數(預設為L2)。
>>> from pyspark.ml.linalg import Vectors
>>> svec = Vectors.sparse(4, {1: 4.0, 3: 3.0})
>>> df = spark.createDataFrame([(Vectors.dense([3.0, -4.0]), svec)], ["dense", "sparse"])
>>> normalizer = Normalizer(p=2.0, inputCol="dense", outputCol="features")
>>> normalizer.transform(df).head().features
DenseVector([0.6, -0.8])
>>> normalizer.setParams(inputCol="sparse", outputCol="freqs").transform(df).head().freqs
SparseVector(4, {1: 0.8, 3: 0.6})
>>> params = {normalizer.p: 1.0, normalizer.inputCol: "dense", normalizer.outputCol: "vector"}
>>> normalizer.transform(df, params).head().vector
DenseVector([0.4286, -0.5714])
>>> normalizerPath = temp_path + "/normalizer"
>>> normalizer.save(normalizerPath)
>>> loadedNormalizer = Normalizer.load(normalizerPath)
>>> loadedNormalizer.getP() == normalizer.getP()
True
pyspark.ml.feature.OneHotEncoder(self, dropLast=True, inputCol=None, outputCol=None)
(分類列編碼為二進位制向量列)
一個熱門的編碼器,將一列類別索引對映到一列二進位制向量,每行至多有一個單值,表示輸入類別索引。 例如,對於5個類別,輸入值2.0將對映到[0.0,0.0,1.0,0.0]的輸出向量。 最後一個類別預設不包含(可通過dropLast進行配置),因為它使向量條目總和為1,因此線性相關。 所以一個4.0的輸入值對映到[0.0,0.0,0.0,0.0]。這與scikit-learn的OneHotEncoder不同,後者保留所有類別。 輸出向量是稀疏的。
用於將分類值轉換為分類索引的StringIndexer.
>>> stringIndexer = StringIndexer(inputCol="label", outputCol="indexed")
>>> model = stringIndexer.fit(stringIndDf)
>>> td = model.transform(stringIndDf)
>>> encoder = OneHotEncoder(inputCol="indexed", outputCol="features")
>>> encoder.transform(td).head().features
SparseVector(2, {0: 1.0})
>>> encoder.setParams(outputCol="freqs").transform(td).head().freqs
SparseVector(2, {0: 1.0})
>>> params = {encoder.dropLast: False, encoder.outputCol: "test"}
>>> encoder.transform(td, params).head().test
SparseVector(3, {0: 1.0})
>>> onehotEncoderPath = temp_path + "/onehot-encoder"
>>> encoder.save(onehotEncoderPath)
>>> loadedEncoder = OneHotEncoder.load(onehotEncoderPath)
>>> loadedEncoder.getDropLast() == encoder.getDropLast()
True
OneHotEncoder可以實現對變數的啞變數化
參考
pyspark.ml.feature.PCA(self, k=None, inputCol=None, outputCol=None)
PCA訓練一個模型將向量投影到前k個主成分的較低維空間。
#-*-coding:utf-8-*-
from pyspark import SparkContext
from pyspark.sql import SparkSession
from pyspark.ml.feature import PCA
from pyspark.ml.feature import PCAModel
from pyspark.ml.linalg import Vectors
if __name__=="__main__":
sc=SparkContext(appName="myApp")
spark=SparkSession.builder.getOrCreate()
data=[(Vectors.sparse(5,[(1,1.0),(3,7.0)]),),
(Vectors.dense([2.0, 0.0, 3.0, 4.0, 5.0]),),
(Vectors.dense([4.0, 0.0, 0.0, 6.0, 7.0]),)]
df=spark.createDataFrame(data,["features"])
"""
df.show()
+--------------------+
| features |
+--------------------+
| (5, [1, 3], [1.0, 7.0]) |
| [2.0, 0.0, 3.0, 4.0, ... |
| [4.0, 0.0, 0.0, 6.0, ... |
+--------------------+
"""
pca=PCA(k=2,inputCol="features",outputCol="pcaFeatures")
model=pca.fit(df)
# print(model.explainedVariance)
# [0.794393253223, 0.205606746777]
pcadf=model.transform(df)
"""
pcadf.show()
+--------------------+--------------------+
| features | pcaFeatures |
+--------------------+--------------------+
| (5, [1, 3], [1.0, 7.0]) | [1.64857282308838... |
| [2.0, 0.0, 3.0, 4.0, ... | [-4.6451043317815... |
| [4.0, 0.0, 0.0, 6.0, ... | [-6.4288805356764... |
+--------------------+--------------------+
"""
temp_path="/tmp/test/"
pca_path="%spca"%(temp_path)
pca.save(pca_path)
pcaload=PCA.load(pca_path)
# print(pcaload.getK()==pca.getK())
# True
pcaModel_path="%spcaModel"%(temp_path)
model.save(pcaModel_path)
modelLoad=PCAModel.load(pcaModel_path)
# print(modelLoad.pc==model.pc)
# True
# print(modelLoad.explainedVariance==model.explainedVariance)
# True
pyspark.ml.feature.QuantileDiscretizer(self, numBuckets=2, inputCol=None, outputCol=None, relativeError=0.001, handleInvalid="error")
與Bucketizer方法類似,但不是傳遞分隔引數,而是傳遞一個numBuckets引數,然後該方法通過計算資料的近似分位數來決定分隔應該是什麼。
>>> values = [(0.1,), (0.4,), (1.2,), (1.5,), (float("nan"),), (float("nan"),)]
>>> df = spark.createDataFrame(values, ["values"])
>>> qds = QuantileDiscretizer(numBuckets=2,
... inputCol="values", outputCol="buckets", relativeError=0.01, handleInvalid="error")
>>> qds.getRelativeError()
0.01
>>> bucketizer = qds.fit(df)
>>> qds.setHandleInvalid("keep").fit(df).transform(df).count()
6
>>> qds.setHandleInvalid("skip").fit(df).transform(df).count()
4
>>> splits = bucketizer.getSplits()
>>> splits[0]
-inf
>>> print("%2.1f" % round(splits[1], 1))
0.4
>>> bucketed = bucketizer.transform(df).head()
>>> bucketed.buckets
0.0
>>> quantileDiscretizerPath = temp_path + "/quantile-discretizer"
>>> qds.save(quantileDiscretizerPath)
>>> loadedQds = QuantileDiscretizer.load(quantileDiscretizerPath)
>>> loadedQds.getNumBuckets() == qds.getNumBuckets()
True
pyspark.ml.feature.StandardScaler(self, withMean=False, withStd=True, inputCol=None, outputCol=None)
(標準化列,使其擁有零均值和等於1的標準差)
通過使用訓練集中樣本的列彙總統計消除平均值和縮放到單位方差來標準化特徵。使用校正後的樣本標準偏差計算“單位標準差”,該標準偏差計算為無偏樣本方差的平方根。
>>> from pyspark.ml.linalg import Vectors
>>> df = spark.createDataFrame([(Vectors.dense([0.0]),), (Vectors.dense([2.0]),)], ["a"])
>>> standardScaler = StandardScaler(inputCol="a", outputCol="scaled")
>>> model = standardScaler.fit(df)
>>> model.mean
DenseVector([1.0])
>>> model.std
DenseVector([1.4142])
>>> model.transform(df).collect()[1].scaled
DenseVector([1.4142])
>>> standardScalerPath = temp_path + "/standard-scaler"
>>> standardScaler.save(standardScalerPath)
>>> loadedStandardScaler = StandardScaler.load(standardScalerPath)
>>> loadedStandardScaler.getWithMean() == standardScaler.getWithMean()
True
>>> loadedStandardScaler.getWithStd() == standardScaler.getWithStd()
True
>>> modelPath = temp_path + "/standard-scaler-model"
>>> model.save(modelPath)
>>> loadedModel = StandardScalerModel.load(modelPath)
>>> loadedModel.std == model.std
True
>>> loadedModel.mean == model.mean
True
pyspark.ml.feature.VectorAssembler(self, inputCols=None, outputCol=None)
非常有用,將多個數字(包括向量)列合併為一列向量
#-*-coding:utf-8-*-
from pyspark import SparkContext
from pyspark.sql import SparkSession
from pyspark.ml.feature import VectorAssembler
if __name__=="__main__":
sc=SparkContext(appName="myApp")
spark=SparkSession.builder.getOrCreate()
df=spark.createDataFrame([(12, 10, 3), (1, 4, 2)],["a","b","c"])
"""
df.show()
+---+---+---+
| a | b | c |
+---+---+---+
| 12 | 10 | 3 |
| 1 | 4 | 2 |
+---+---+---+
"""
vectorAssembler=VectorAssembler(inputCols=["a","b","c"],outputCol="features")
vdf=vectorAssembler.transform(df)
"""
vdf.show()
+---+---+---+---------------+
| a | b | c | features |
+---+---+---+---------------+
| 12 | 10 | 3 | [12.0, 10.0, 3.0] |
| 1 | 4 | 2 | [1.0, 4.0, 2.0] |
+---+---+---+---------------+
"""
temp_path="/tmp/test/"
vectorAssembler_path="%svectorAssembler"
vectorAssembler.save(vectorAssembler_path)
vectorAssemblerLoad=VectorAssembler.load(vectorAssembler_path)
# print(vectorAssemblerLoad.transform(df).head().features==vectorAssembler.transform(df).head().features)
# True
-
pyspark.ml.feature.VectorIndexer(self, maxCategories=20, inputCol=None, outputCol=None)類別列生成索引向量 -
pyspark.ml.feature.VectorSlicer(self, inputCol=None, outputCol=None, indices=None, names=None)作用於特徵向量,給定一個索引列表,從特徵向量中提取值。
特徵變換–標籤和索引的轉化
來源:阮榕城 2017年12月18日
在機器學習處理過程中,為了方便相關演算法的實現,經常需要把標籤資料(一般是字串)轉化成整數索引,或是在計算結束後將整數索引還原為相應的標籤。
Spark ML包中提供了幾個相關的轉換器,例如:StringIndexer,IndexToString,OneHotEncoder,VectorIndexer,它們提供了十分方便的特徵轉換功能,這些轉換器類都位於pyspark.ml.feature包下。
值得注意的是,用於特徵轉換的轉換器和其他的機器學習演算法一樣,也屬於ML Pipeline模型的一部分,可以用來構成機器學習流水線,以StringIndexer為例,其儲存著進行標籤數值化過程的相關 超引數,是一個Estimator,對其呼叫fit(…)方法即可生成相應的模型StringIndexerModel類,很顯然,它儲存了用於DataFrame進行相關處理的 引數,是一個Transformer(其他轉換器也是同一原理)
下面對幾個常用的轉換器依次進行介紹。
StringIndexer
StringIndexer轉換器可以把一列類別型的特徵(或標籤)進行編碼,使其數值化,索引的範圍從0開始,該過程可以使得相應的特徵索引化,使得某些無法接受類別型特徵的演算法可以使用,並提高諸如決策樹等機器學習演算法的效率。
索引構建的順序為標籤的頻率,優先編碼頻率較大的標籤,所以出現頻率最高的標籤為0號。
如果輸入的是數值型的,我們會把它轉化成字元型,然後再對其進行編碼。
首先,引入必要的包,並建立一個簡單的DataFrame,它只包含一個id列和一個標籤列category:
from pyspark.ml.feature import StringIndexer
df = spark.createDataFrame([(0, "a"), (1, "b"), (2, "c"), (3, "a"), (4, "a"), (5, "c")],["id", "category"])
隨後,我們建立一個StringIndexer物件,設定輸入輸出列名,其餘引數採用預設值,並對這個DataFrame進行訓練,產生StringIndexerModel物件:
indexer = StringIndexer(inputCol="category", outputCol="categoryIndex")
model = indexer.fit(df)
隨後即可利用該物件對DataFrame進行轉換操作,可以看到,StringIndexerModel依次按照出現頻率的高低,把字元標籤進行了排序,即出現最多的“a”被編號成0,“c”為1,出現最少的“b”為0。
indexed = model.transform(df)
indexed.show()
+---+--------+-------------+
| id|category|categoryIndex|
+---+--------+-------------+
| 0| a| 0.0|
| 1| b| 2.0|
| 2| c| 1.0|
| 3| a| 0.0|
| 4| a| 0.0|
| 5| c| 1.0|
+---+--------+-------------+
IndexToString
與StringIndexer相對應,IndexToString的作用是把標籤索引的一列重新映射回原有的字元型標籤。
其主要使用場景一般都是和StringIndexer配合,先用StringIndexer將標籤轉化成標籤索引,進行模型訓練,然後在預測標籤的時候再把標籤索引轉化成原有的字元標籤。當然,你也可以另外定義其他的標籤。
首先,和StringIndexer的實驗相同,我們用StringIndexer讀取資料集中的“category”列,把字元型標籤轉化成標籤索引,然後輸出到“categoryIndex”列上,構建出新的DataFrame。
from pyspark.ml.feature import IndexToString, StringIndexer
df = spark.createDataFrame([(0, "a"), (1, "b"), (2, "c"), (3, "a"), (4, "a"), (5, "c")],["id", "category"])
indexer = StringIndexer(inputCol="category", outputCol="categoryIndex")
model = indexer.fit(df)
indexed = model.transform(df)
隨後,建立IndexToString物件,讀取“categoryIndex”上的標籤索引,獲得原有資料集的字元型標籤,然後再輸出到“originalCategory”列上。最後,通過輸出“originalCategory”列,可以看到資料集中原有的字元標籤。
converter = IndexToString(inputCol="categoryIndex", outputCol="originalCategory")
converted = converter.transform(indexed)
converted.show()
"""
+---+--------+-------------+----------------+
| id|category|categoryIndex|originalCategory|
+---+--------+-------------+----------------+
| 0| a| 0.0| a|
| 1| b| 2.0| b|
| 2| c| 1.0| c|
| 3| a| 0.0| a|
| 4| a| 0.0| a|
| 5| c| 1.0| c|
+---+--------+-------------+----------------+
"""
converted.select("id", "categoryIndex", "originalCategory").show()
+---+-------------+----------------+
| id|categoryIndex|originalCategory|
+---+-------------+----------------+
| 0| 0.0| a|
| 1| 2.0| b|
| 2| 1.0| c|
| 3| 0.0| a|
| 4| 0.0| a|
| 5| 1.0| c|
+---+-------------+----------------+
可以看到,並沒有用到原來的model也能變換回來,說明傳入的indexed裡面保留了原來的對映關係,這樣才能映射回去。
在sklearn中是通過le = LabelEncoder(),le.fit(),然後le就可以去做對映le.transform(),最後再通過le.inverse_transform(yPred)映射回來。
OneHotEncoder
獨熱編碼(One-Hot Encoding) 是指把一列類別性特徵(或稱名詞性特徵,nominal/categorical features)對映成一系列的二元連續特徵的過程,原有的類別性特徵有幾種可能取值,這一特徵就會被對映成幾個二元連續特徵,每一個特徵代表一種取值,若該樣本表現出該特徵,則取1,否則取0。
One-Hot編碼適合一些期望類別特徵為連續特徵的演算法,比如說邏輯斯蒂迴歸等。
首先建立一個DataFrame,其包含一列類別性特徵,需要注意的是,在使用OneHotEncoder進行轉換前,DataFrame需要先使用StringIndexer將原始標籤數值化:
from pyspark.ml.feature import OneHotEncoder, StringIndexer
df = spark.createDataFrame([
(0, "a"),
(1, "b"),
(2, "c"),
(3, "a"),
(4, "a"),
(5, "c")
], ["id", "category"])
stringIndexer = StringIndexer(inputCol="category", outputCol="categoryIndex")
model = stringIndexer.fit(df)
indexed = model.transform(df)
隨後,我們建立OneHotEncoder物件對處理後的DataFrame進行編碼,可以看見,編碼後的二進位制特徵呈稀疏向量形式,與StringIndexer編碼的順序相同,需注意的是最後一個Category(”b”)被編碼為全0向量,若希望”b”也佔有一個二進位制特徵,則可在建立OneHotEncoder時指定setDropLast(false)。
encoder = OneHotEncoder(inputCol="categoryIndex", outputCol="categoryVec")
encoded = encoder.transform(indexed)
encoded.show()
+---+--------+-------------+-------------+
| id|category|categoryIndex| categoryVec|
+---+--------+-------------+-------------+
| 0| a| 0.0|(2,[0],[1.0])|
| 1| b| 2.0| (2,[],[])|
| 2| c| 1.0|(2,[1],[1.0])|
| 3| a| 0.0|(2,[0],[1.0])|
| 4| a| 0.0|(2,[0],[1.0])|
| 5| c| 1.0|(2,[1],[1.0])|
+---+--------+-------------+-------------+
VectorIndexer
之前介紹的StringIndexer是針對單個類別型特徵進行轉換,倘若所有特徵都已經被組織在一個向量中,又想對其中某些單個分量進行處理時,Spark ML提供了VectorIndexer類來解決向量資料集中的類別性特徵轉換。
通過為其提供maxCategories超引數,它可以自動識別哪些特徵是類別型的,並且將原始值轉換為類別索引。它基於不同特徵值的數量來識別哪些特徵需要被類別化,那些取值可能性最多不超過maxCategories的特徵需要會被認為是類別型的。
在下面的例子中,我們讀入一個數據集,然後使用VectorIndexer訓練出模型,來決定哪些特徵需要被作為類別特徵,將類別特徵轉換為索引,這裡設定maxCategories為10,即只有種類小10的特徵才被認為是類別型特徵,否則被認為是連續型特徵:
from pyspark.ml.feature import VectorIndexer
data = spark.read.format('libsvm').load('file:///usr/local/spark/data/mllib/sample_libsvm_data.txt')
indexer = VectorIndexer(inputCol="features", outputCol="indexed", maxCategories=10)
indexerModel = indexer.fit(data)
categoricalFeatures = indexerModel.categoryMaps
indexedData = indexerModel.transform(data)
indexedData.show()
+-----+--------------------+--------------------+
|label| features| indexed|
+-----+--------------------+--------------------+
| 0.0|(692,[127,128,129...|(692,[127,128,129...|
| 1.0|(692,[158,159,160...|(692,[158,159,160...|
| 1.0|(692,[124,125,126...|(692,[124,125,126...|
| 1.0|(692,[152,153,154...|(692,[152,153,154...|
| 1.0|(692,[151,152,153...|(692,[151,152,153...|
| 0.0|(692,[129,130,131...|(692,[129,130,131...|
| 1.0|(692,[158,159,160...|(692,[158,159,160...|
| 1.0|(692,[99,100,101,...|(692,[99,100,101,...|
| 0.0|(692,[154,155,156...|(692,[154,155,156...|
| 0.0|(692,[127,128,129...|(692,[127,128,129...|
| 1.0|(692,[154,155,156...|(692,[154,155,156...|
| 0.0|(692,[153,154,155...|(692,[153,154,155...|
| 0.0|(692,[151,152,153...|(692,[151,152,153...|
| 1.0|(692,[129,130,131...|(692,[129,130,131...|
| 0.0|(692,[154,155,156...|(692,[154,155,156...|
| 1.0|(692,[150,151,152...|(692,[150,151,152...|
| 0.0|(692,[124,125,126...|(692,[124,125,126...|
| 0.0|(692,[152,153,154...|(692,[152,153,154...|
| 1.0|(692,[97,98,99,12...|(692,[97,98,99,12...|
| 1.0|(692,[124,125,126...|(692,[124,125,126...|
+-----+--------------------+--------------------+
Spark ML 幾種 歸一化(規範化)方法總結
4種不同的歸一化方法:
- Normalizer
- StandardScaler
- MinMaxScaler
- MaxAbsScaler
Normalizer
Normalizer的作用範圍是每一行,使每一個行向量的範數變換為一個單位範數,下面的示例程式碼都來自spark官方文件加上少量改寫和註釋。
// L^1 norm將每一行的規整為1階範數為1的向量,1階範數即所有值絕對值之和。
+---+--------------+------------------+
| id| features| normFeatures|
+---+--------------+------------------+
| 0|[1.0,0.5,-1.0]| [0.4,0.2,-0.4]|
| 1| [2.0,1.0,1.0]| [0.5,0.25,0.25]|
| 2|[4.0,10.0,2.0]|[0.25,0.625,0.125]|
+---+--------------+------------------+
// L^inf norm向量的無窮階範數即向量中所有值中的最大值
+---+--------------+--------------+
| id| features| normFeatures|
+---+--------------+--------------+
| 0|[1.0,0.5,-1.0]|[1.0,0.5,-1.0]|
| 1| [2.0,1.0,1.0]| [1.0,0.5,0.5]|
| 2|[4.0,10.0,2.0]| [0.4,1.0,0.2]|
+---+--------------+--------------+
StandardScaler
StandardScaler處理的物件是每一列,也就是每一維特徵,將特徵標準化為單位標準差或是0均值,或是0均值單位標準差。
主要有兩個引數可以設定:
- withStd: 預設為真。將資料標準化到單位標準差。
- withMean: 預設為假。是否變換為0均值。 (此種方法將產出一個稠密輸出,所以不適用於稀疏輸入。)
StandardScaler需要fit資料,獲取每一維的均值和標準差,來縮放每一維特徵。
StandardScaler是一個Estimator,它可以fit資料集產生一個StandardScalerModel,用來計算彙總統計。
然後產生的模可以用來轉換向量至統一的標準差以及(或者)零均值特徵。
注意如果特徵的標準差為零,則該特徵在向量中返回的預設值為0.0。
// 將每一列的標準差縮放到1。
+---+--------------+------------------------------------------------------------+
|id |features |scaledFeatures |
+---+--------------+------------------------------------------------------------+
|0 |[1.0,0.5,-1.0]|[0.6546536707079772,0.09352195295828244,-0.6546536707079771]|
|1 |[2.0,1.0,1.0] |[1.3093073414159544,0.1870439059165649,0.6546536707079771] |
|2 |[4.0,10.0,2.0]|[2.618614682831909,1.870439059165649,1.3093073414159542] |
+---+--------------+------------------------------------------------------------+
MinMaxScaler
MinMaxScaler作用同樣是每一列,即每一維特徵。將每一維特徵線性地對映到指定的區間,通常是[0, 1]。
MinMaxScaler計算資料集的彙總統計量,併產生一個MinMaxScalerModel。
注意因為零值轉換後可能變為非零值,所以即便為稀疏輸入,輸出也可能為稠密向量。
該模型可以將獨立的特徵的值轉換到指定的範圍內。
它也有兩個引數可以設定:
- min: 預設為0。指定區間的下限。
- max: 預設為1。指定區間的上限。
// 每維特徵線性地對映,最小值對映到0,最大值對映到1。
+--------------+-----------------------------------------------------------+
|features |scaledFeatures |
+--------------+-----------------------------------------------------------+
|[1.0,0.5,-1.0]|[0.0,0.0,0.0] |
|[2.0,1.0,1.0] |[0.3333333333333333,0.05263157894736842,0.6666666666666666]|
|[4.0,10.0,2.0]|[1.0,1.0,1.0] |
+--------------+-----------------------------------------------------------+
MaxAbsScaler
MaxAbsScaler將每一維的特徵變換到[-1, 1]閉區間上,通過除以每一維特徵上的最大的絕對值,它不會平移整個分佈,也不會破壞原來每一個特徵向量的稀疏性。
因為它不會轉移/集中資料,所以不會破壞資料的稀疏性。
// 每一維的絕對值的最大值為[4, 10, 2]
+--------------+----------------+
| features| scaledFeatures|
+--------------+----------------+
|[1.0,0.5,-1.0]|[0.25,0.05,-0.5]|
| [2.0,1.0,1.0]| [0.5,0.1,0.5]|
|[4.0,10.0,2.0]| [1.0,1.0,1.0]|
+--------------+----------------+
ml中的資料統計特性
描述性統計
mllib中有count()、max()、min()、mean()、 normL1()、normL2() 、numNonzeros() 、 variance()等基本的統計功能,不知道為什麼在ml中沒有了。
但是可以利用pysparl.sql.DataFrame來處理相關的統計
df.agg(*exprs)計算均值、最大值等df.approxQuantile(col, probabilities, relativeError)計算分位數df.corr(col1, col2, method=None)計算相關係數df.describe(*cols)計算統計特性
>>> df.describe().show()
+-------+------------------+-----+
|summary| age| name|
+-------+------------------+-----+
| count| 2| 2|
| mean| 3.5| null|
| stddev|2.1213203435596424| null|
| min| 2|Alice|
| max| 5| Bob|
+-------+------------------+-----+
df.summary(*statistics)
>>> df.summary().show()
+-------+------------------+-----+
|summary| age| name|
+-------+------------------+-----+
| count| 2| 2|
| mean| 3.5| null|
| stddev|2.1213203435596424| null|
| min| 2|Alice|
| 25%| 2| null|
| 50%| 2| null|
| 75%| 5| null|
| max| 5| Bob|
+-------+------------------+-----+
>>> df.summary("count", "min", "25%", "75%", "max").show()
+-------+---+-----+
|summary|age| name|
+-------+---+-----+
| count| 2| 2|
| min| 2|Alice|
| 25%| 2| null|
| 75%| 5| null|
| max| 5| Bob|
+-------+---+-----+
df.dropna(how='any', thresh=None, subset=None)df.fillna(value, subset=None)df.avg(*cols)count()max(*cols)mean(*cols)min(*cols)pivot(pivot_col, values=None)sum(*cols)
相關性
class pyspark.ml.stat.Correlation
#-*-coding:utf-8-*-
from pyspark import SparkContext
from pyspark.sql import SparkSession
from pyspark.ml.linalg import Vectors
from pyspark.ml.stat import Correlation
if __name__=="__main__":
sc=SparkContext(appName="myApp")
spark=SparkSession.builder.getOrCreate()
dataset=[
[Vectors.dense([1, 0, 0, -2])],
[Vectors.dense([4, 5, 0, 3])],
[Vectors.dense([6, 7, 0, 8])],
[Vectors.dense([9, 0, 0, 1])]
]
df=spark.createDataFrame(dataset,["features"])
dfCorr=Correlation.corr(dataset=df,column="features",method="pearson")
"""
dfCorr.show()
+--------------------+
| pearson(features)|
+--------------------+
|1.0 ...|
+--------------------+
print(dfCorr.collect())
[Row(pearson(features) = DenseMatrix(4, 4, [1.0, 0.0556, nan, 0.4005, 0.0556, 1.0, nan, 0.9136, nan, nan, 1.0, nan,
0.4005, 0.9136, nan, 1.0], False))]
"""
dfCorrelation=dfCorr.collect()[0][0]
"""
print(print(str(dfCorrelation).replace('nan', 'NaN')))
DenseMatrix([[ 1. , 0.05564149, NaN, 0.40047142],
[ 0.05564149, 1. , NaN, 0.91359586],
[ NaN, NaN, 1. , NaN],
[ 0.40047142, 0.91359586, NaN, 1. ]])
"""
統計檢驗
class pyspark.ml.stat.ChiSquareTest
#-*-coding:utf-8-*-
from pyspark import SparkContext
from pyspark.sql import SparkSession
from pyspark.ml.linalg import Vectors
from pyspark.ml.stat import ChiSquareTest
if __name__=="__main__":
sc=SparkContext(appName="myApp")
spark=SparkSession.builder.getOrCreate()
dataset=[
[0,Vectors.dense([0, 0, 1])],
[0,Vectors.dense([1, 0, 1])],
[1,Vectors.dense([2, 1, 1])],
[1,Vectors.dense([3, 1, 1])]
]
df=spark.createDataFrame(dataset,["label","features"])
dfChiSq=ChiSquareTest.test(df,featuresCol="features",labelCol="label")
"""
dfChiSq.show()
+--------------------+----------------+-------------+
| pValues|degreesOfFreedom| statistics|
+--------------------+----------------+-------------+
|[0.26146412994911...| [3, 1, 0]|[4.0,4.0,0.0]|
+--------------------+----------------+-------------+
"""