【程式碼】K-means聚類詳解及實現 (Matlab聚類工具箱和自己實現)
一. 聚類
先說說聚類。顧名思義,就是有一團資料,根據某種準則把相似的資料分別聚在一起,形成不同的類別(每個類別稱為一簇)。聚類是一種無監督的演算法。所謂無監督就是說,雖然聚類把物體分類到了不同的簇,只能知道哪些資料是屬於同一類的,至於這一類資料到底是什麼,並不知道。
聚類分為原型聚類、系統聚類、密度聚類。
原型聚類:先找幾個原型(隨機找k個元素作為k個類的中心),其他元素加入到距離最近的類裡去,再次計算中心,根據距離重新分類,至中心不變為止。
系統聚類:每個元素各成一類,然後根據類間的距離,合併距離最小的,直至一類。
密度聚類:(等學了再補充)
對於距離:
樣本之間常採用閔科夫斯基距離(minkowski),歐式距離等。
類間的距離有最短距離(single)、最長距離(complex)、中間距離(median)、重心距離(centriod)、平均距離(average)、離差平方和距離(ward)等。
聚類效果評價 (https://cn.mathworks.com/help/stats/cluster-evaluation.html matlab提供了相關函式包,相關準則參考matlab的工具箱支援)
1. Calinski Harabasz Evaluation[1]2.Davies Bouldin Criterion [2]3.Gap Value [3]4. Silhouette criterion [4]
二. K-means
下面這篇文章寫的挺好的,可以參考。
http://blog.csdn.net/mingtian715/article/details/51534165
三.程式碼
下面放一段自己寫的matlab程式碼,不使用for迴圈,適合大量資料處理。復刻了周志華機器學習204頁k-means給西瓜分類的結果。
%2017/10/27 K-means
%ref. 周志華 西瓜分類
clc;
clear;
close all;
data = [0.697 0.460;0.774,0.376;0.634,0.264;0.608,0.318;0.556,0.215;0.403,0.237;
0.481,0.149;0.437,0.211;0.666,0.091;0.243,0.267;0.245,0.057;0.343,0.099;
0.639 0.161;0.657,0.198;0.360,0.370;0.593,0.042;0.719,0.103;0.359,0.188;
0.339,0.241;0.282,0.257;0.748,0.232;0.714,0.346;0.483,0.312;0.478,0.437;
0.525,0.369;0.751,0.489;0.532,0.472;0.473,0.376;0.725,0.445;0.446,0.459;]
%聚類 數值初始化
% scatter(data(:,1),data(:,2))
% axis([0.1 0.9 0 0.8])
num = length(data); %樣本數
fea_n=2; %特徵數
k = 3; %簇數
%如果各特徵數值差異較大,需要歸一化
% maxd = max(data,2);
% data(:,1:fea_n)=data(:,1:fea_n)./repmat(maxd(1,1:fea_n),num,1);
% totalMean = mean(data(:,1:fea_n));
%產生不相等隨機數
% b = randperm(num);
% r = b(1:k);
r = [6 12 24];%書中隨機選取的初始化質心
%初始均值向量 u 1行 * k*fea_n列
u = [data(r(1,1),1:fea_n)];
for n = 2:k
u = [u , data(r(1,n),1:fea_n)];
end
count = 1;%迭代次數
flag = 1;
cellshape([1:k],1) = fea_n;
while(flag&count<=4)
if count == 1 %第迭代迴圈初始化
old_u = u;
end
difer = (repmat(data(:,1:fea_n),1,k) - repmat(old_u,num,1));
cell = mat2cell((difer.*difer),[num],cellshape); %按簇劃分子塊
distcell = cell2mat(cellfun(@(x) sum(x,2).^0.5, cell,'UniformOutput',false)'); %計算距離
dist=reshape(distcell,[],k);
[mini,lambda] = min(dist,[],2); %找到最近似的矩陣
data = [data(:,1:fea_n),lambda]; %標記資料
data = sortrows(data,3);
%各簇內記錄的數量
for n = 1:k
len(1,n) = length(find(lambda==n));
end
datacell = mat2cell(data(:,1:fea_n)',[fea_n],len);
new_u = cell2mat(cellfun(@(x) mean(x,2),datacell,'UniformOutput',false)')';
flag = length(find(new_u-old_u~=0));
old_u = new_u;
count=count+1;
end
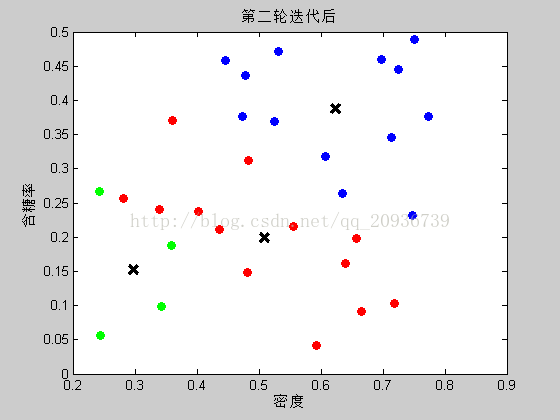
%畫出分類結果
Color_map = hsv(k);
Label = data(:,fea_n+1);
Color_Label = Color_map(Label,:);
figure(1),clf
C=reshape(new_u',2,[])';
plot(C(:,1),C(:,2),'kx','MarkerSize',10,'LineWidth',3)
hold on;
scatter(data(:,1),data(:,2),40,Color_Label,'filled')
xlabel('密度')
ylabel('含糖率')
title('第四輪迭代後')
四.結果、
五.聚類效果評價
用matlab自帶的工具箱進行聚類效果分析,
rng('default'); % For reproducibility
eva = evalclusters(data,'kmeans','CalinskiHarabasz','KList',[1:10])
plot(eva)
評價結果
eva =
CalinskiHarabaszEvaluation (帶屬性):
NumObservations: 30
InspectedK: [1 2 3 4 5 6 7 8 9 10]
CriterionValues: [NaN 22.9791 28.0930 35.4862 33.0436 32.7692 29.9349 33.5191 32.8026 34.0822]
OptimalK: 4
k=4是比較合適的,根據CH指標越大越好的準則,k=4有最好的結果。
引用
[1] Calinski, T., and J. Harabasz. “A dendrite method for cluster analysis.” Communications in Statistics. Vol. 3, No. 1, 1974, pp. 1–27.
[2] Davies, D. L., and D. W. Bouldin. “A Cluster Separation Measure.” IEEE Transactions on Pattern Analysis and Machine Intelligence. Vol. PAMI-1, No. 2, 1979, pp. 224–227.
[3] Tibshirani, R., G. Walther, and T. Hastie. “Estimating the number of clusters in a data set via the gap statistic.” Journal of the Royal Statistical Society: Series B. Vol. 63, Part 2, 2001, pp. 411–423.