IMex和IntAct資料庫簡介
阿新 • • 發佈:2018-11-08
歡迎關注微信公眾號《生信修煉手冊》!
蛋白質相互作用的資料庫非常的多,比如DIP, MINT, IntAct, BioGRID等,不同資料庫中的資訊存在了大量的冗餘,而且在不同資料庫之間進行檢索也非常的費力,為了減少不同資料庫的冗餘,最大化提升資料儲存和檢索的效率,由多個數據庫的開發團隊和維護者共同參與成立了一個委員會,名字叫做international molecular exchange consortium, 簡稱IMEx, 該委員會致力於將不同資料庫中的資訊合併,減少冗餘並提供了一個統一的查詢工具
該委員會的成員示意如下


該專案的地址如下
http://www.imexconsortium.org/
通過官網的檢索框,可以方便的檢索各個成員資料庫中的內容,示意如下

以tp53為例,檢索結果如下

點選IMEx的連結,可以看到如下所示的表格
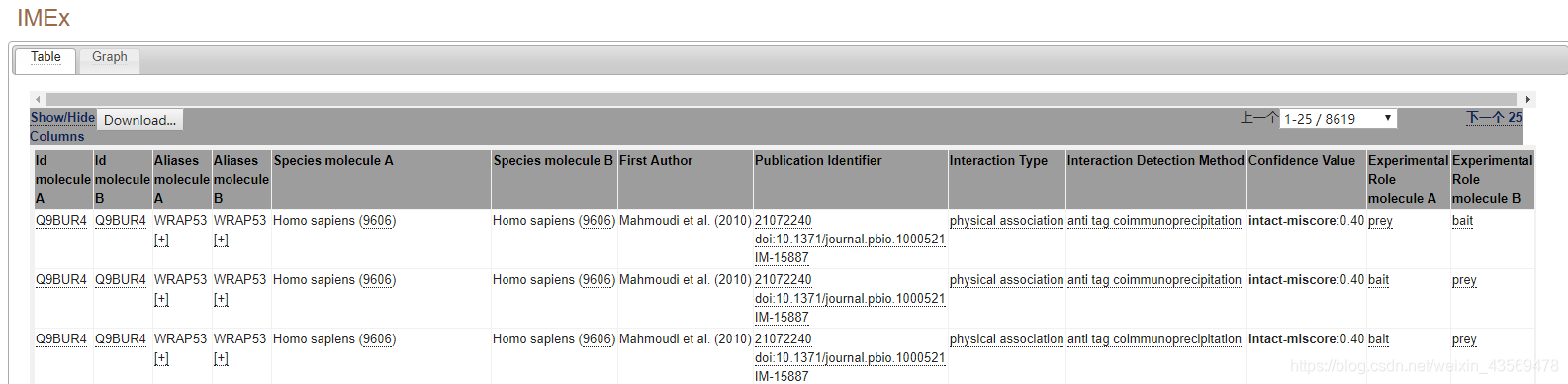
採用了MITAB格式來展示蛋白質相互作用資訊,通過左上角的Download按鈕可以進行下載,通過IMEx的官網,我們可以方便的查詢某個蛋白的相關作用網路資訊的,但是下載資料並不是非常的方便,如果要下載資料,還是要藉助IntAct資料庫。IntAct資料庫是IMEx的成員資料庫之一,目前該資料也整合了IMEx的所有資料,網址如下
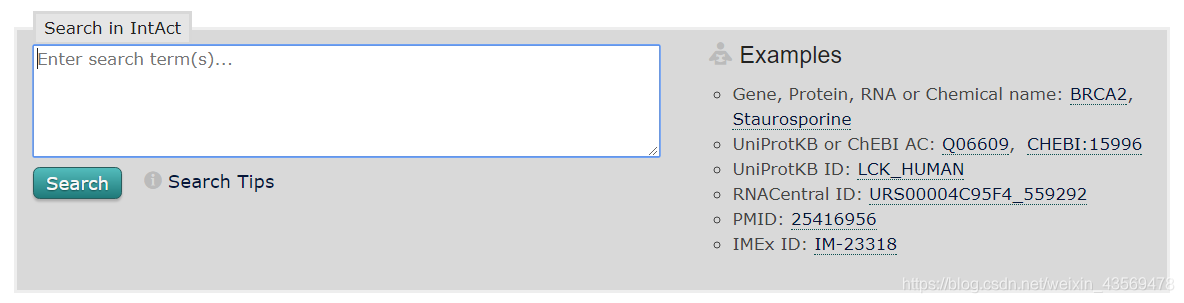
線上檢索功能肯定是有的,如下所示,支援多種查詢方式
在檢索結果的頁面,可以自定義展示的列資訊,也可以選擇對應的格式進行下載,示意如下
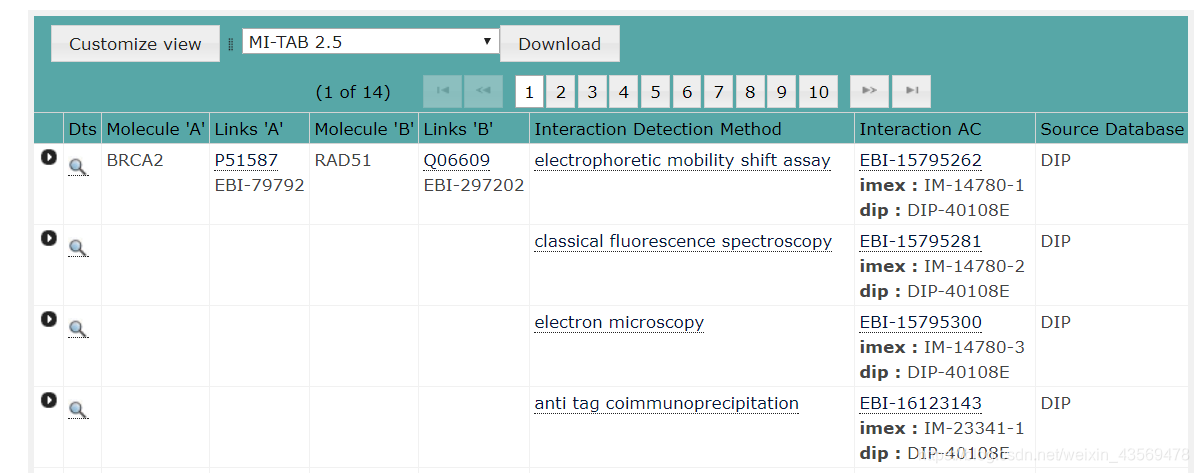
通過
Graph按鈕,還可以顯示查詢蛋白的相互作用網路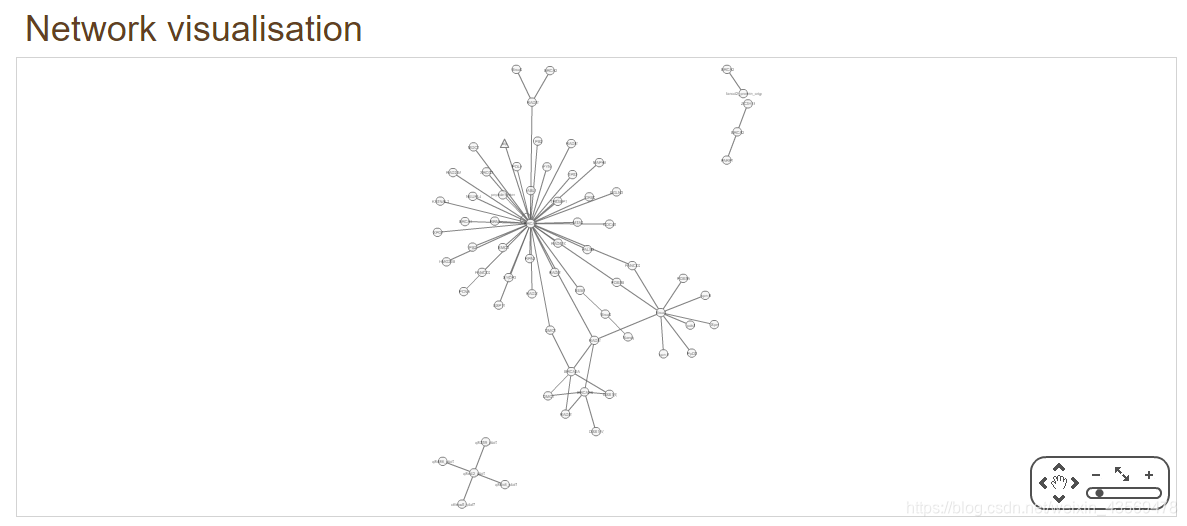
和string資料庫相比,IntAct的檢索功能也有其侷限性,只適用於查詢單個蛋白的相互作用網路,無法查詢輸入的多個蛋白之間的相互作用網路。如果要查詢多個蛋白之間的相互作用網路,只能自己寫指令碼從整個相互作用網路中提取資訊,官網提供了下載功能,示意如下

提供了多種格式的下載,主要有
XML和TAB兩種格式, PSI-MI格式就是xml格式的檔案,PSI-MI TAB就是\t分隔的檔案,TAB格式的內容相對直觀,而XML格式適用於程式處理。
對於蛋白質相互作用網路而言,String資料庫的使用更加簡單,更加的人性化,所以知名度更高,而IMEx和IntAct的使用有一定的技術門檻,但是可以作為String資料庫的一種補充,能夠得到更加豐富的資訊。
掃描關注微訊號,更多精彩內容等著你!

