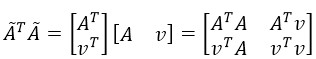稀疏編碼中的正交匹配追蹤(OMP)與程式碼
分享一下我老師大神的人工智慧教程!零基礎,通俗易懂!http://blog.csdn.net/jiangjunshow
也歡迎大家轉載本篇文章。分享知識,造福人民,實現我們中華民族偉大復興!
最近在看有關匹配追蹤與相關優化的文章,發現了這篇http://blog.csdn.net/scucj/article/details/7467955,感覺作者寫得很不錯,這裡也再寫寫自己的理解。文中有Matlab的程式碼,為了方便以後的使用,我順便寫了一個C++版本的,方便與OpenCV配合。
為了方便理解,我將所有向量都表示為平面二維向量,待用原子表徵的目標向量y,用紅色表示,原子向量用藍色表示,殘差向量用綠色表示。於是匹配追蹤演算法(MP)實際上可以用下圖表示。

注意原子向量和目標向量都已歸一化到單位長度,MP演算法首先在所有原子向量中找到向OA投影最大的向量,即OB,然後計算OA - <OA, OB>OB,其中的<OA, OB>OB也就是圖中的OD了,被OA減掉後,剩下的就是殘差DA,根據初中幾何知識就可以知道,DA是一定垂直於OB的,也就是說MP的殘差始終與最近選出來的那個原子向量正交。
而OMP要做的,就是讓殘差與已經選出來的所有原子向量都正交,這一點在圖上不好畫出來,但上面的那篇博文寫的已經很詳盡了,這裡不再敖述。
下面是用C++實現的OMP演算法,具體流程參考上面博文中的一張圖:

其中的最小二乘可以直接通過矩陣運算得到,也可以使用OpenCV的solve方法,該方法專門用於求解線性方程組或最小二乘問題。程式碼如下:
void OrthMatchPursuit( vector<Mat>& dic,//字典 Mat& target, float min_residual, int 程式碼寫得有點亂,基本上完全按照演算法步驟來的,應該還有很大的效能提升空間。
===================================無恥的分割線========================================
之前說上面的程式碼還有很大的優化空間,這幾天搗鼓了一下,發現優化還是很有成效的,下面是具體方法。
為方便起見,這裡用A代表從字典當中選出的原子的集合,對於上面求解最小二乘的一步,可以表示為下式:
其中的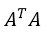
那麼用這個新的原子集合計算x時,可以得到:
可以看到,新的集合乘積,有一部分是上次的結果(上式最右邊矩陣的第一個元素),因此沒有必要每次都從新計算,而只需對原來的矩陣更新一列和一行就行了。同時,上式的第二和第三個元素互為轉置,也只需要計算其中一個,第四個元素是v的二範數的平方,直接呼叫norm()函式求得。
在對矩陣進行行和列的新增時,我放棄了使用vconcat和hconcat方法,這兩個方法效率較低,每次新增都會把原來的部分複製一遍。我現在一次分配好需要的大小,然後通過Mat的括號操作符取需要的子矩陣進行更新和計算。
求解x時,還有一步是求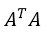
我做了一個測試,使用一個有600多個原子的字典,每個原子的維度為200,稀疏度設定為10,匹配一個訊號,原來的方法需要200ms左右,而用上面的方法優化後,只需10ms!!快了一個數量級!!
下面是優化後的程式碼:
void DictionaryLearning::OrthMatchPursuit( Mat& target, float min_residual, int sparsity, //Store matched patches' coefficient vector<float>& coefficients, //Store matched patches vector<DicPatch>& matched_patches, //Store indices of matched patches vector<int>& matched_indices ){ Mat residual = target.clone(); //the atoms' set; Mat ori_phi = Mat::zeros(m_vec_dims,sparsity,CV_32FC1); Mat phi; //phi.t()*phi which is a SPD matrix Mat ori_spd = Mat::ones(sparsity,sparsity,CV_32FC1); Mat spd = ori_spd(Rect(0,0,1,1)); //reserve enough memory. matched_patches.reserve(sparsity); matched_indices.reserve(sparsity); float max_coefficient; int matched_index; deque<DicPatch>::iterator matched_patch_it; for(int spars = 1;;spars++) { max_coefficient = 0; matched_index = 0; int current_index = 0; for (deque<DicPatch>::iterator patch_it = m_patches.begin(); patch_it != m_patches.end(); ++patch_it ) { Mat& cur_vec = (*patch_it).vector; float coefficient = (float)cur_vec.dot(residual); //Find the maxmum coefficient if (abs(coefficient) > abs(max_coefficient)) { max_coefficient = coefficient; matched_patch_it = patch_it; matched_index = current_index; } current_index++; } matched_patches.push_back((*matched_patch_it)); matched_indices.push_back(matched_index); Mat& matched_vec = (*matched_patch_it).vector; //update the spd matrix via symply appending a single row and column to it. if (spars > 1) { Mat v = matched_vec.t()*phi; float c = (float)norm(matched_vec); Mat new_row = ori_spd(Rect(0, spars - 1, spars - 1, 1)); Mat new_col = ori_spd(Rect(spars - 1, 0, 1, spars - 1)); v.copyTo(new_row); ((Mat)v.t()).copyTo(new_col); *ori_spd.ptr<float>(spars - 1, spars - 1) = c*c; spd = ori_spd(Rect(0, 0, spars, spars)); } //Add the new matched patch to the vectors' set. phi = ori_phi(Rect(0, 0, spars, m_vec_dims)); matched_vec.copyTo(phi.col(spars - 1)); //A SPD matrix! Use Cholesky process to speed up. Mat x = spd.inv(DECOMP_CHOLESKY)*phi.t()*target; residual = target - phi*x; float res_norm = (float)norm(residual); if (spars >= sparsity || res_norm <= min_residual) { coefficients.clear(); coefficients.reserve(x.cols); x.copyTo(coefficients); return; } }}給我老師的人工智慧教程打call!http://blog.csdn.net/jiangjunshow

最近在看有關匹配追蹤與相關優化的文章,發現了這篇http://blog.csdn.net/scucj/article/details/7467955,感覺作者寫得很不錯,這裡也再寫寫自己的理解。文中有Matlab的程式碼,為了方便以後的使用,我順便寫了一個C++版本的,方便與OpenCV配合。
為了方便理解,我將所有向量都表示為平面二維向量,待用原子表徵的目標向量y,用紅色表示,原子向量用藍色表示,殘差向量用綠色表示。於是匹配追蹤演算法(MP)實際上可以用下圖表示。

注意原子向量和目標向量都已歸一化到單位長度,MP演算法首先在所有原子向量中找到向OA投影最大的向量,即OB,然後計算OA - <OA, OB>OB,其中的<OA, OB>OB也就是圖中的OD了,被OA減掉後,剩下的就是殘差DA,根據初中幾何知識就可以知道,DA是一定垂直於OB的,也就是說MP的殘差始終與最近選出來的那個原子向量正交。
而OMP要做的,就是讓殘差與已經選出來的所有原子向量都正交,這一點在圖上不好畫出來,但上面的那篇博文寫的已經很詳盡了,這裡不再敖述。
下面是用C++實現的OMP演算法,具體流程參考上面博文中的一張圖:

其中的最小二乘可以直接通過矩陣運算得到,也可以使用OpenCV的solve方法,該方法專門用於求解線性方程組或最小二乘問題。程式碼如下:
void OrthMatchPursuit( vector<Mat>& dic,//字典 Mat& target, float min_residual, int sparsity, Mat& x, //返回每個原子對應的係數; vector<int>& patch_indices //返回選出的原子序號 ){ Mat residual = target.clone(); Mat phi; //儲存已選出的原子向量 x.create(0, 1, CV_32FC1); float max_coefficient; unsigned int patch_index; for(;;) { max_coefficient = 0; for (int i = 0; i < dic.size(); i++) { float coefficient = (float)dic[i].dot(residual); if (abs(coefficient) > abs(max_coefficient)) { max_coefficient = coefficient; patch_index = i; } } patch_indices.push_back(patch_index); //新增選出的原子序號 Mat& matched_patch = dic[patch_index]; if (phi.cols == 0) phi = matched_patch; else hconcat(phi,matched_patch,phi); //將新原子合併到原子集合中(都是列向量) x.push_back(0.0f); //對係數矩陣新加一項 solve(phi, target, x, DECOMP_SVD); //求解最小二乘問題 residual = target - phi*x; //更新殘差 float res_norm = (float)norm(residual); if (x.rows >= sparsity || res_norm <= min_residual) //如果殘差小於閾值或達到要求的稀疏度,就返回 return; }}程式碼寫得有點亂,基本上完全按照演算法步驟來的,應該還有很大的效能提升空間。
===================================無恥的分割線========================================
之前說上面的程式碼還有很大的優化空間,這幾天搗鼓了一下,發現優化還是很有成效的,下面是具體方法。
為方便起見,這裡用A代表從字典當中選出的原子的集合,對於上面求解最小二乘的一步,可以表示為下式:
其中的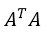
那麼用這個新的原子集合計算x時,可以得到:
可以看到,新的集合乘積,有一部分是上次的結果(上式最右邊矩陣的第一個元素),因此沒有必要每次都從新計算,而只需對原來的矩陣更新一列和一行就行了。同時,上式的第二和第三個元素互為轉置,也只需要計算其中一個,第四個元素是v的二範數的平方,直接呼叫norm()函式求得。
在對矩陣進行行和列的新增時,我放棄了使用vconcat和hconcat方法,這兩個方法效率較低,每次新增都會把原來的部分複製一遍。我現在一次分配好需要的大小,然後通過Mat的括號操作符取需要的子矩陣進行更新和計算。
求解x時,還有一步是求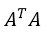
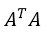
我做了一個測試,使用一個有600多個原子的字典,每個原子的維度為200,稀疏度設定為10,匹配一個訊號,原來的方法需要200ms左右,而用上面的方法優化後,只需10ms!!快了一個數量級!!
下面是優化後的程式碼:
void DictionaryLearning::OrthMatchPursuit( Mat& target, float min_residual, int sparsity, //Store matched patches' coefficient vector<float>& coefficients, //Store matched patches vector<DicPatch>& matched_patches, //Store indices of matched patches vector<int>& matched_indices ){ Mat residual = target.clone(); //the atoms' set; Mat ori_phi = Mat::zeros(m_vec_dims,sparsity,CV_32FC1); Mat phi; //phi.t()*phi which is a SPD matrix Mat ori_spd = Mat::ones(sparsity,sparsity,CV_32FC1); Mat spd = ori_spd(Rect(0,0,1,1)); //reserve enough memory. matched_patches.reserve(sparsity); matched_indices.reserve(sparsity); float max_coefficient; int matched_index; deque<DicPatch>::iterator matched_patch_it; for(int spars = 1;;spars++) { max_coefficient = 0; matched_index = 0; int current_index = 0; for (deque<DicPatch>::iterator patch_it = m_patches.begin(); patch_it != m_patches.end(); ++patch_it ) { Mat& cur_vec = (*patch_it).vector; float coefficient = (float)cur_vec.dot(residual); //Find the maxmum coefficient if (abs(coefficient) > abs(max_coefficient)) { max_coefficient = coefficient; matched_patch_it = patch_it; matched_index = current_index; } current_index++; } matched_patches.push_back((*matched_patch_it)); matched_indices.push_back(matched_index); Mat& matched_vec = (*matched_patch_it).vector; //update the spd matrix via symply appending a single row and column to it. if (spars > 1) { Mat v = matched_vec.t()*phi; float c = (float)norm(matched_vec); Mat new_row = ori_spd(Rect(0, spars - 1, spars - 1, 1)); Mat new_col = ori_spd(Rect(spars - 1, 0, 1, spars - 1)); v.copyTo(new_row); ((Mat)v.t()).copyTo(new_col); *ori_spd.ptr<float>(spars - 1, spars - 1) = c*c; spd = ori_spd(Rect(0, 0, spars, spars)); } //Add the new matched patch to the vectors' set. phi = ori_phi(Rect(0, 0, spars, m_vec_dims)); matched_vec.copyTo(phi.col(spars - 1)); //A SPD matrix! Use Cholesky process to speed up. Mat x = spd.inv(DECOMP_CHOLESKY)*phi.t()*target; residual = target - phi*x; float res_norm = (float)norm(residual); if (spars >= sparsity || res_norm <= min_residual) { coefficients.clear(); coefficients.reserve(x.cols); x.copyTo(coefficients); return; } }}