R中雙表操作學習[轉載]
阿新 • • 發佈:2018-12-27
轉自:https://www.jianshu.com/p/a7af4f6e50c3
1.原始資料
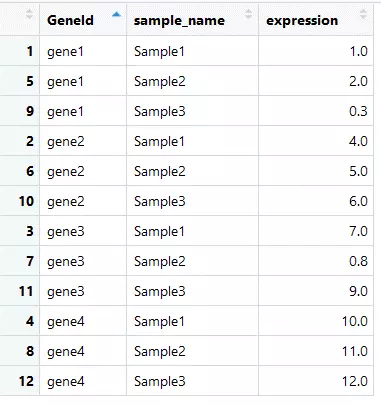
以上是原有的一個,再生成一個新的:
> gene_exp_tidy2 <- data.frame(GeneId = rep("gene5", times = 3),sample_name = paste("sample", 1:3, sep = ""), expression = 2:4) > gene_exp_tidy2 GeneId sample_name expression 1 gene5 sample1 2 2gene5 sample2 3 3 gene5 sample3 4 > mode(gene_exp_tidy2) [1] "list" > typeof(gene_exp_tidy2) [1] "list"
//這個生成資料框的一句程式碼好厲害啊,學習了。
2.資料框拼接
# 1.使用R自帶的函式rbind拼接 rbind(gene_exp_tidy,gene_exp_tidy2) # 2.使用dplyr的bind_rows函式 bind_rows(gene_exp_tidy,gene_exp_tidy2)
其中對dplyr中拼接函式的說明:

包括按行合併和按列合併。也就是會改變表中y的值。
3.交集
3.1構建一個gene1和gene5組成的資料框
gene_exp_tidy3 <- filter(gene_exp_tidy, GeneId == "gene1")%>% bind_rows(gene_exp_tidy2)
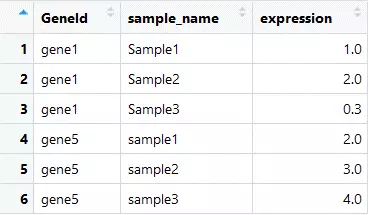
//就是過濾吧,從原來的當中根據列名過濾,列名中的內容=gene1的列,然後進行合併到變數2的行上。結果如上。
3.2求交集
# 求兩個資料框的交集 intersect(gene_exp_tidy, gene_exp_tidy3)

3.3並集(自動去重)
# 求兩個資料框的並集(自動去除重複)
union(gene_exp_tidy, gene_exp_tidy3)

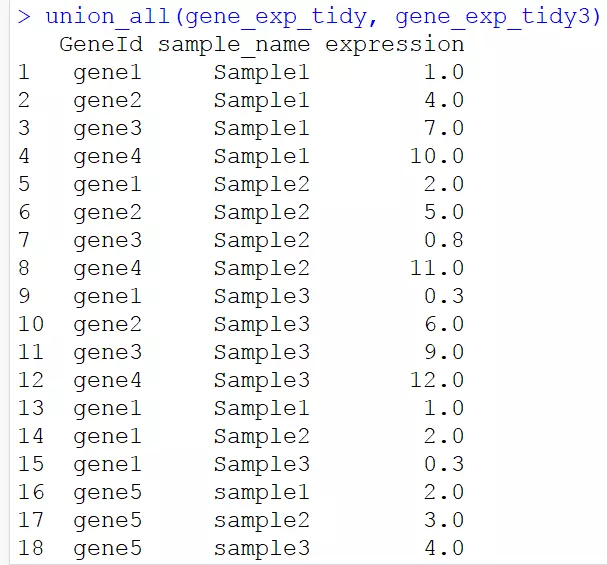
3.3並集(不去重)
# 求兩個資料框的並集,不去除重複
union_all(gene_exp_tidy, gene_exp_tidy3)
//是三個元素完全相同,才會判斷為相同的。
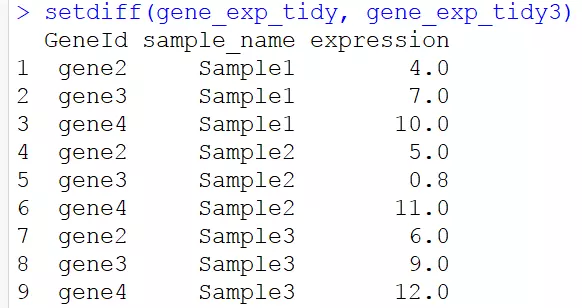
3.4求差集
# 求兩個資料框的差集
setdiff(gene_exp_tidy, gene_exp_tidy3)
4表連線操作
4.1內連線
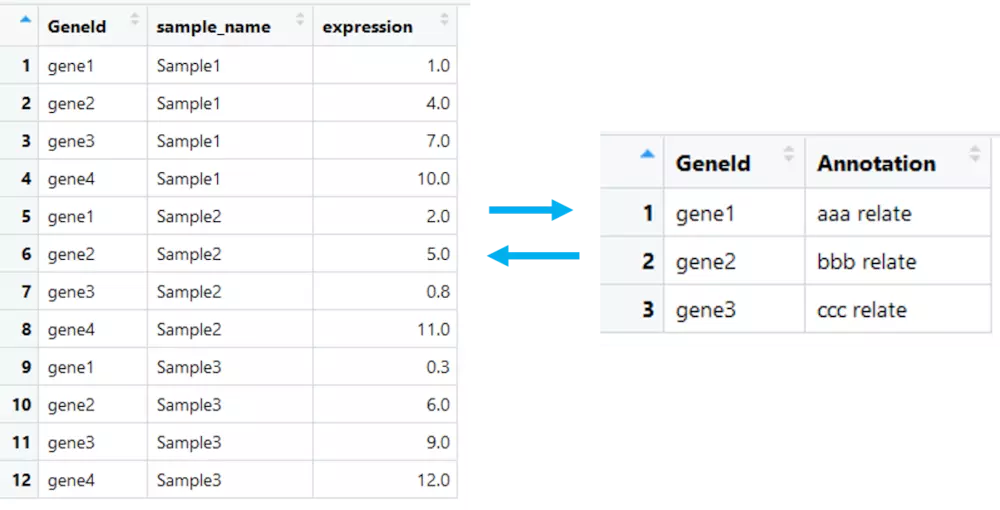
上圖中,左邊的資料框中有基因的表達資訊,右邊的資料框中有基因的功能註釋,如何把兩個資料框相關聯,將基因功能註釋新增到基因的表達資訊中呢?
//也就是以左邊的表為基準,檢視當前geneid是否在右邊表中出現,如果出現,則合併唄。
左連線:以第一個資料框為準,將第二個資料框的相關資訊新增進去
# 將兩張表以GeneId進行關聯 left_join(gene_exp_tidy, gene_anno, by = "GeneId")
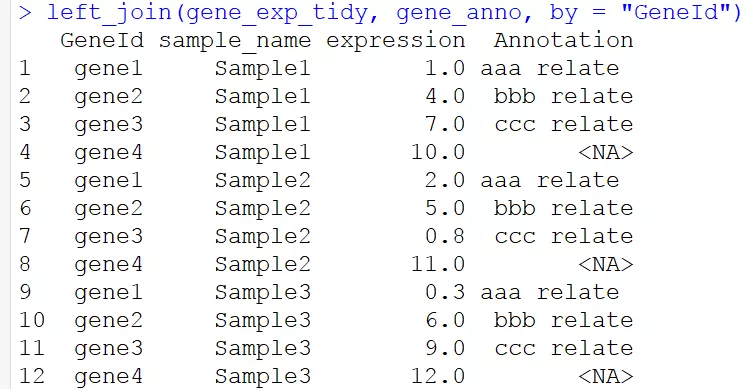
對於如果兩個表的列名不恰好相同的話,使用以下語句:
# 使用rename函式將GeneId換成Geneid gene_anno <- rename(gene_anno, Geneid = GeneId) # 將兩個資料框關聯,設定兩個資料框中相同的列名 left_join(gene_exp_tidy, gene_anno, by =c("GeneId" = "Geneid"))
by中是一個向量,左邊表的列名=右邊表的列名 。結果與上述是一樣的結果。
4.2右連線
右連線:以第二個資料框為準,將第一個資料框中的相關資訊新增進去
# 右連線 right_join(gene_exp_tidy, gene_anno, by =c("GeneId" = "Geneid"))

4.3內連線:保留兩個資料框中共有的GeneId
# 內連線 inner_join(gene_exp_tidy, gene_anno, by =c("GeneId" = "Geneid"))

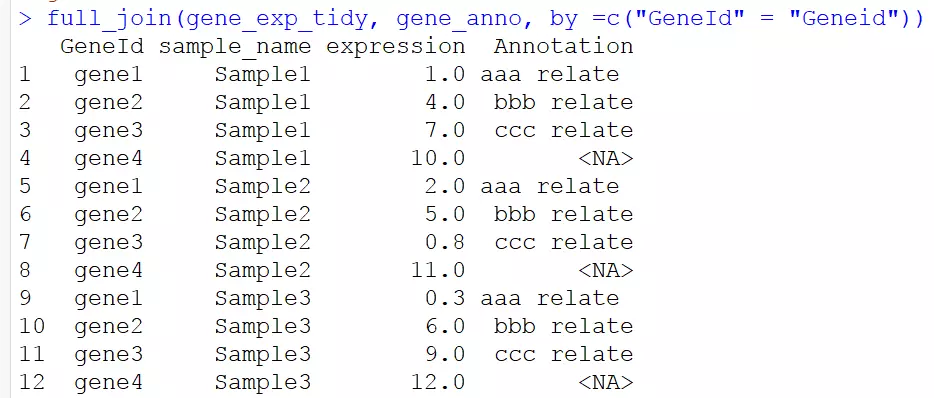
4.4全連線:保留兩張表格中所有GeneId的資訊
# 全連線 full_join(gene_exp_tidy, gene_anno, by =c("GeneId" = "Geneid"))
4.5半連線
半連線:只保留第二個資料框中包含的GeneId資訊
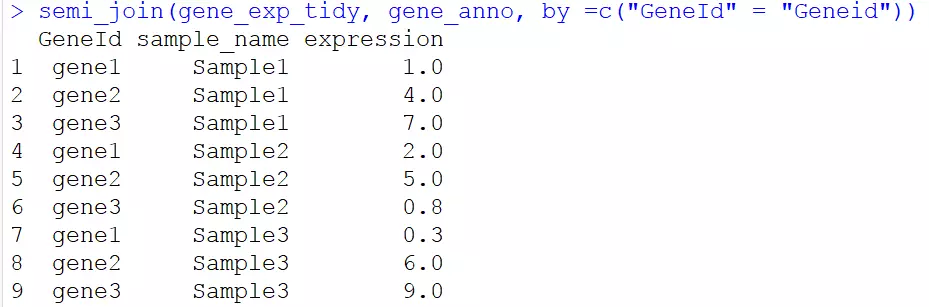
//應該是隻根據c中的保留表2中的列,不會進行其他列的資訊合併。
4.6反連線:只保留第二個資料框中不包含的GeneId資訊
# 反連線 anti_join(gene_exp_tidy, gene_anno, by =c("GeneId" = "Geneid"))

