基因序列分類問題——多類分類器的設計
阿新 • • 發佈:2019-01-08
1. 問題背景
生物學研究中,用檢測DNA序列來判斷檢測物件的物種越來越成為一種簡便、快捷的手段。現在的測量技術已經能夠很方便地測量出物種的DNA序列,將其進行分類可以運用機器學習的方法。本文將探討如何基於DNA序列的資料集設計一個性能良好的多類的分類器。
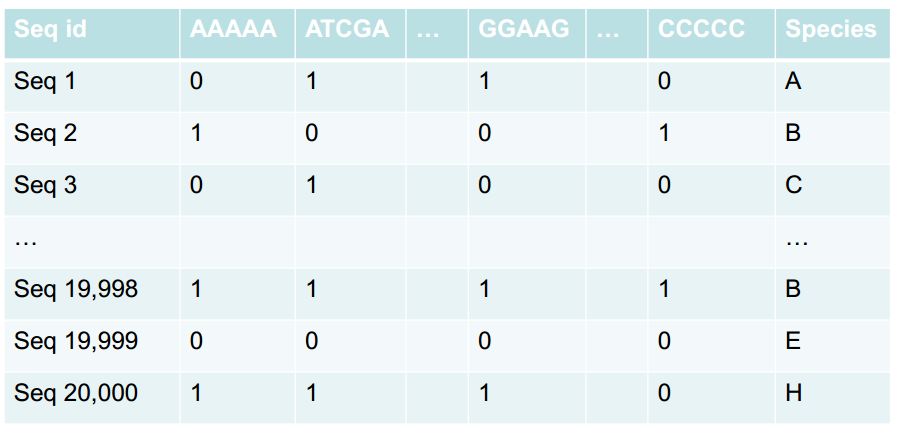
2. 資料集簡介
資料集中有共20000條基於序列,一共來自10個物種。其中一條基因是一個72個鹼基的序列,如下所示:
AGGGGGCTGGCCCGTGACGAGCGGACCATCGTCGGCACCCCCGAGACGATCGCCGACCACATCCAGGAGTGG在分類器的設計過程中,可以將資料集分割成訓練集和測試集。如採用5-fold交叉驗證時,將資料集均分成5份,每次取其中一份作為測試集,其餘四份作為訓練集,將五次的準確率取平均作為分類器的準確率。
3. 資料處理和特徵提取
基因序列這樣的原始資料不能直接用於計算,因此我們將對資料進修處理得到特徵。
設K=5,則特徵是長度為5的基因片段,總共有4^5個。
AAAAA, AAAAT, AAAAG, AAAAC,
AAATA …
ATCGA…TTTTT…GGGGG…CCCCC每個特徵即基因片段的值是基因序列中該基因片段出現的次數。
4. 演算法設計
我們先回顧下整個問題,現在要設計一個10類的分類器,如下圖所示。

最簡單的分類演算法是二類logistical regression,然而現在我們要處理的是多類問題,因此要採用 Multinomial logistic regression,也稱softmax演算法。
另外SVM分類演算法也是非常出名的,它高效,效能好,被很多人認為是分類演算法中最好的。
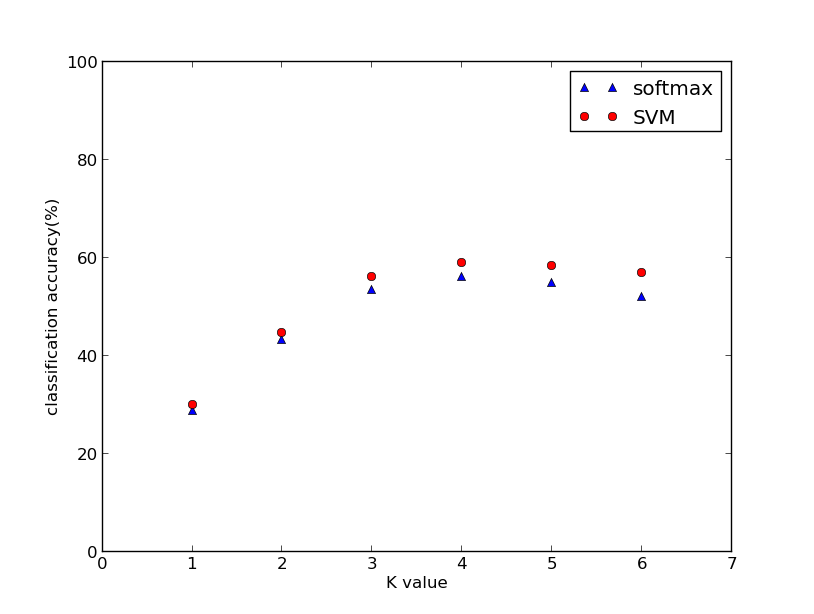
5. 結果分析
K值選取對分類的影響
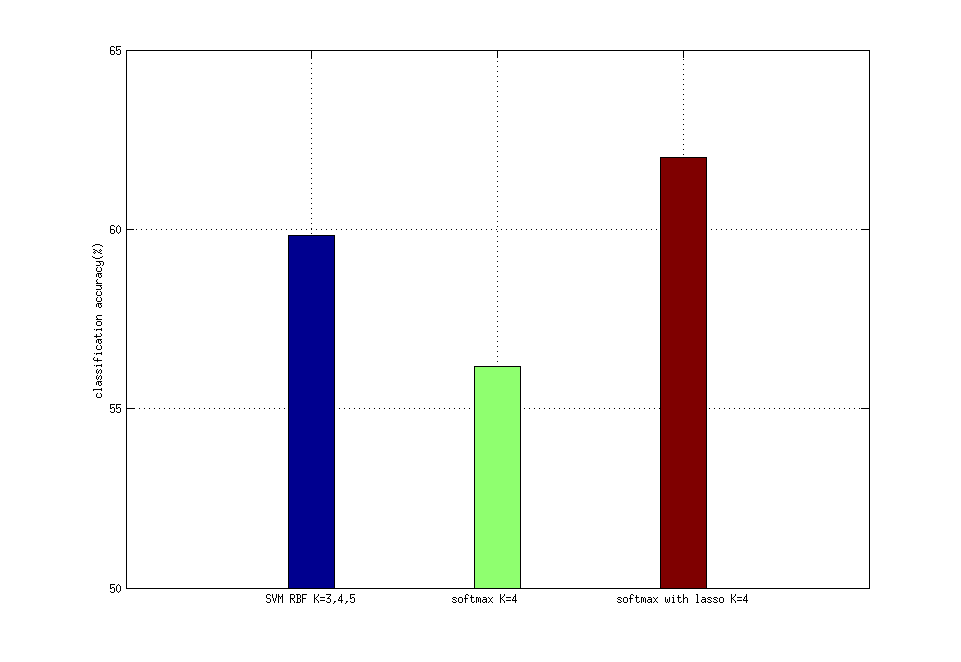
兩種演算法的對比
6. 提高與增強
為了進一步提高準確率,再做一些嘗試。
6.1引入更多特徵
綜合K=3,4,5的特徵,但是這樣又使得特徵的維度太大,可能混入了太多不顯著的特徵使得準確率不會提高,因此輔以PCA降維。實驗結果如下。
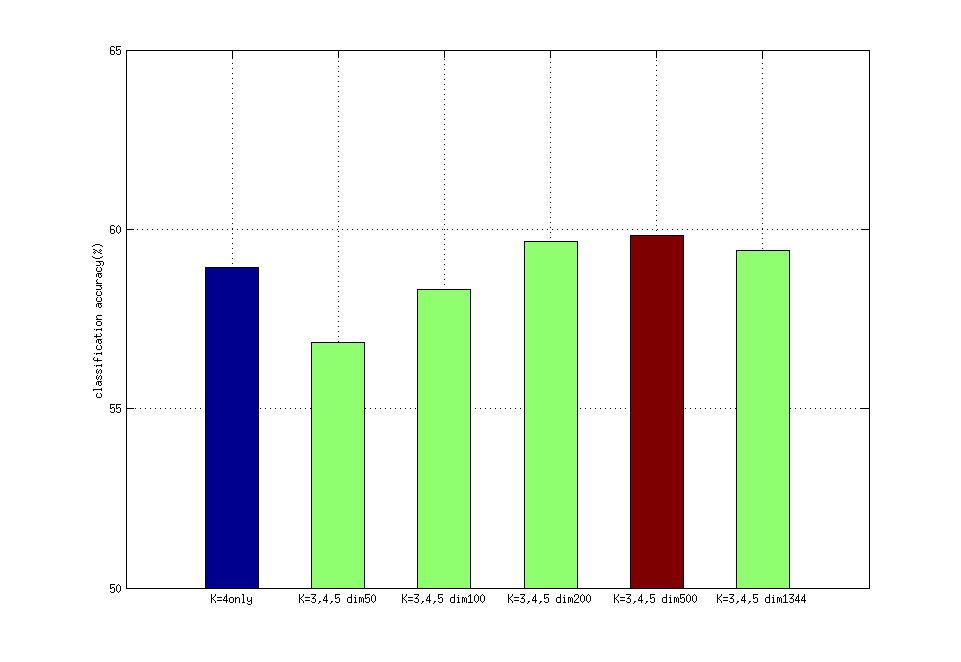
可以看到K=3,4,5,dim=500時,準確率比K=4時有所提高。
6.2使用lasso進行特徵的篩選
lasso演算法可以篩選出顯著的特徵,以降低無關特徵對分類的影響。先通過lasso篩選特徵,再用softmax構建分類器,我們得到了本文中最好的分類器,準確率高達63%,大大超過了不使用特徵選取的softmax和最好的SVM分類器。可見下圖。