MATLAB影象處理實驗——細胞影象的分割和計數
1. 實驗內容
本實驗為使用MATLAB編寫的細胞影象分割及計數系統,實現了對影象內細胞的計數,以及對每個細胞周長和麵積的測量,並分別展示了分割後的每個細胞的影象。
實驗步驟共分為影象預處理、影象預分割、空洞填充、黏連細胞分割、細胞個數統計、細胞特徵統計及顯示。
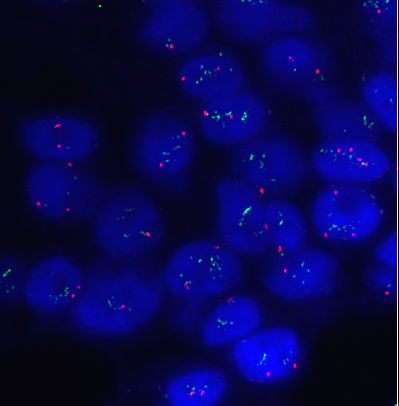
實驗所用影象如下圖所示:
2. 影象預處理
使用濾波對細胞影象進行保邊去噪的處理,這步的主要作用是去除影象噪聲,系統中提供了中值濾波、均值濾波、維納濾波、銳化濾波,經過測試發現中值濾波的效果最好,因此以中值濾波為例進行下面的實驗。
部分程式碼如下:
global original_img show_img; str=get(hObject,'string'); axes(handles.axes1); [m,n,k]=size(original_img); switch str case '原圖' show_img=original_img; imshow(show_img); case '中值濾波' show_img=original_img; for i=1:3 show_img(:,:,i)=medfilt2(show_img(:,:,i)); end imshow(show_img); case '均值濾波' show_img=original_img; h=ones(3,3)/9; for i=1:3 show_img(:,:,i)=imfilter(show_img(:,:,i),h); end imshow(show_img); case '維納濾波' show_img=original_img; for i=1:3 show_img(:,:,i)=wiener2(show_img(:,:,i),[5 5]); end imshow(show_img); case '銳化濾波' show_img=original_img; h=[0 1 0;1 -4 1;0 1 0]; for i=1:3 J=conv2(im2double(show_img(:,:,i)),h,'same'); show_img(:,:,i)=show_img(:,:,i)-uint8(J); end imshow(show_img); end
中值濾波影象如下:
3. 預分割
選擇彩色細胞影象的藍色通道,並對該通道的灰度圖,用大津法Otsu進行二值化預分割,將細胞作為前景分割出來。
global show_img;
B=show_img(:,:,3);
B_gray=im2double(B);
level=graythresh(B_gray);
B_bw=im2bw(B_gray,level);
axes(handles.axes1);
show_img=B_bw;
imshow(show_img);預分割影象如下:
4. 孔洞填充
對分割後的二值圖,實施開運算,填充細胞中的孔洞,使輕微粘連細胞分開及細小的細胞消失。
global show_img;
se=strel('disk',5);
Spot_filled=imopen(show_img,se);
axes(handles.axes1);
show_img=Spot_filled;
imshow(show_img);填充孔洞後的影象如下:
5. 黏連細胞分割
使用分水嶺分割,將影象中粘連嚴重的細胞分割開來,對於仍難以分離的細胞,使用手動分割,用滑鼠作為筆,將劃過的畫素點的值設為0,滑鼠鬆開後,再進行一次分水嶺分割。
分水嶺分割程式碼:
global show_img; D=-bwdist(~show_img); mask=imextendedmin(D,2); D2=imimposemin(D,mask); Ld=watershed(D2); Water_splited=show_img; Water_splited(Ld==0)=0; axes(handles.axes1); show_img=Water_splited; imshow(show_img);
手動分割程式碼:
global ButtonDown show_img;
if ButtonDown == 1
pos = get(handles.axes1, 'CurrentPoint');
row=floor(pos(1,2));
col=floor(pos(1,1));
Diy_splited=show_img;
Diy_splited(row,col)=0;
show_img=Diy_splited;
axes(handles.axes1);
imshow(show_img);
end 分割後的影象如下:
6. 細胞個數統計及顯示
首先刪除掉邊緣上的細胞,再利用四連通區域標記演算法統計分割後非粘連細胞的個數,最後在原圖上標記出分割好的細胞,並標號。
細胞個數統計程式碼:
global N show_img original_img img_group perimeter_group area_group;
[B,L,N]=bwboundaries(show_img,'noholes');
[m,n]=size(show_img);
counted_img=zeros(m,n);
for i=1:N
%顯示邊緣
for j=1:size(B{i},1)
counted_img(B{i}(j,1),B{i}(j,2))=1;
end
%標號
med_row=floor(mean(B{i}(:,1)));
med_col=floor(mean(B{i}(:,2)));
if floor(i/10)==0
str=[num2str(i) '.png'];
I=imread(str);
else
str1=[num2str(floor(i/10)) '.png'];
str2=[num2str(rem(i,10)) '.png'];
I1=imread(str1);
I2=imread(str2);
I=[I1 I2];
end
[number_row,number_col]=size(I);
counted_img((med_row - (number_row/2)):(med_row + (number_row/2) - 1),(med_col - (number_col/2)):(med_col + (number_col/2) - 1))=I;
%擷取每個細胞影象
item_img=original_img(med_row - 45:med_row +44,med_col - 45:med_col + 44,:);
item_img_index=L(med_row - 45:med_row +44,med_col - 45:med_col + 44);
index=find(item_img_index~=i);
r=item_img(:,:,1);
g=item_img(:,:,2);
b=item_img(:,:,3);
r(index)=255;
g(index)=255;
b(index)=255;
item_img=cat(3,r,g,b);
img_group{i}=item_img;
%每個細胞周長
item_perimeter=size(B{i},1);
perimeter_group{i}=item_perimeter;
%每個細胞面積
item_area=length(find(L==i));
area_group{i}=item_area;
end原圖細胞標號程式碼:
R=original_img(:,:,1)+uint8(counted_img*255);
G=original_img(:,:,2)+uint8(counted_img*255);
B=original_img(:,:,3)+uint8(counted_img*255);
show_img=cat(3,R,G,B);
axes(handles.axes1);
imshow(show_img);統計結果圖如下:
7. 細胞特徵統計與顯示
對每個細胞,統計其面積,並用提取細胞邊緣,統計邊緣輪廓上的畫素個數即細胞周長,將每個細胞的影象及相關資訊分別顯示出來。
global N img_group perimeter_group area_group page pages;
page=1;
pages=ceil(N/6);
str=['第' num2str(page) '/' num2str(pages) '頁'];
set(handles.page_num,'string',str);
if page~=pages
axes(handles.item1);imshow(img_group{1});
axes(handles.item2);imshow(img_group{2});
axes(handles.item3);imshow(img_group{3});
axes(handles.item4);imshow(img_group{4});
axes(handles.item5);imshow(img_group{5});
axes(handles.item6);imshow(img_group{6});
for i=1:6
strs{i}=['No.' num2str(i) ' ' '周長:' num2str(perimeter_group{i}) ' ' '面積:' num2str(area_group{i})];
end
set(handles.detail_info1,'string',strs{1});
set(handles.detail_info2,'string',strs{2});
set(handles.detail_info3,'string',strs{3});
set(handles.detail_info4,'string',strs{4});
set(handles.detail_info5,'string',strs{5});
set(handles.detail_info6,'string',strs{6});
else
item_img=ones(size(img_group{1}));
item_img(:,:,:)=255;
item_imgs{1:6}=item_img;
item_strs{1:6}='';
for i=1:N
item_imgs{i}=img_group{i};
item_strs{i}=['No.' num2str(i) ' ' '周長:' num2str(perimeter_group{i}) ' ' '面積:' num2str(area_group{i})];
end
axes(handles.item1);imshow(item_imgs{1});
axes(handles.item2);imshow(item_imgs{2});
axes(handles.item3);imshow(item_imgs{3});
axes(handles.item4);imshow(item_imgs{4});
axes(handles.item5);imshow(item_imgs{5});
axes(handles.item6);imshow(item_imgs{6});
set(handles.detail_info1,'string',item_strs{1});
set(handles.detail_info2,'string',item_strs{2});
set(handles.detail_info3,'string',item_strs{3});
set(handles.detail_info4,'string',item_strs{4});
set(handles.detail_info5,'string',item_strs{5});
set(handles.detail_info6,'string',item_strs{6});
end
分頁顯示結果如下:
8. 實驗心得
本次實驗耗時3天完成,實現了實驗要求的所有功能,但由於時間倉促,仍有不少細節地方需要完善。在實驗過程中,有一些功能在程式碼實現上比較有難度,所幸最終找到了解決辦法,以下是我認為在編碼實現過程中遇到的最有難度的三個問題及詳細解決辦法。
(1)手動分割
使用分水嶺分割後,仍有細胞沒有被分割開,需要進行手動分割。方法為通過GUI滑鼠響應,當滑鼠按下和移動時,獲取當前游標所在畫素的橫縱座標,將影象中對應位置處的數值改為0,再顯示新的影象。在滑鼠移動過程中,對影象的修改速度遠遠小於滑鼠滑動速度,這樣只會在滑鼠劃過的路徑上面留下一些黑點,而不是一條線。在滑鼠鬆開時,對影象再進行一次分水嶺分割,就會沿著黑點的路徑生成一條分割線,從而實現對黏連嚴重細胞的手動分割。
(2)原圖細胞標號
首先通過繪圖軟體作出0到9共10個數字的影象,使用MATLAB處理得到每個數字的二值圖用於之後的標號。通過bwboundaries對影象進行標記後,得到每個細胞的邊緣畫素點的橫縱座標,再計算得出每個細胞的中心位置座標,將該座標與對應數字影象的中心座標對應,將數字影象加到原圖上,從而實現對原圖細胞的標號。對於標號是兩位數的細胞,需首先將十位上的數字和各位上的數字拼接,生成新的影象用於標號。
(3)結果分頁顯示
由於對GUI物件的操作無法通過迴圈結構實現,因此首先生成6張全白的影象,再根據這一頁上要顯示的細胞個數,對相應的影象進行修改,最後將這6張影象全部顯示出來,是細胞影象的就顯示細胞影象,不是細胞影象的則顯示全白的影象。