scikit-learn使用PCA降維小結
本文在主成分分析(PCA)原理總結和用scikit-learn學習主成分分析(PCA)的內容基礎上做了一些筆記和補充,強調了我認為重要的部分,其中一些細節不再贅述。
Jupiter notebook版本參見我的github: https://github.com/konatasick/machine_learning_note/blob/master/pca.ipynb
PCA的思想
PCA(Principal components analysis,主成分分析)是一種降維演算法,它通過使樣本間方差儘量大來儘可能保留原始資料的相關關係。
PCA的演算法
1) 對所有的樣本進行中心化 2) 計算樣本的協方差矩陣 3) 對協方差矩陣進行特徵值分解 4)取出最大的m個特徵值對應的特徵向量, 將所有的特徵向量標準化後,組成特徵向量矩陣W。 5)對樣本集中的每一個樣本轉化為新的樣本
scikit-learn的sklearn.decomposition.PCA引數介紹
官方文件:sklearn.decomposition.PCA
Parameters:
n_components:這個代表了需要降維的維度。當它是整數時,代表了保留的維度數量。當它是一個介於0~1之間的數時,代表了主成分的方差所佔的最小比例,例如0.95代表取超過95%的量的維度。當它為‘mle’,同時svd_solver == ‘full’時,系統會根據MLE演算法自動選擇維度。(此時svd_solver == ‘auto’將會被解讀為svd_solver == ‘full’)
svd_solver:預設是‘auto’,即在剩下的‘full’, ‘arpack’, ‘randomized’中根據情況選一個。‘full’是傳統的PCA,‘arpack’, ‘randomized’適用於資料量大的場景,其區別在於前者是通過scipy.sparse.linalg.svds實現。
Attributes*:
components_ : 主成分的投影座標,代表了資料的最大方差的方向,根據explainedvariance由大到小排列。維度是m*n,其中n是原始資料的維度,m是降維後的維度。
explainedvariance
explained_varianceratio=explained_variance/sum(explainedvariance)
維度是m, 當m=n時,sum(explained_varianceratio)=1。
mean_:每個feature的平均值。在pca演算法的第一步,需要對feature歸一化,此時的平均值保留在這裡。
ncomponents :模型實際的降維數,即m。
PCA例項
用scikit-learn學習主成分分析(PCA)中示範了降維的操作。
首先我們生成隨機資料並可視化,程式碼如下:
import numpy as np import matplotlib.pyplot as plt from mpl_toolkits.mplot3d import Axes3D %matplotlib inline from sklearn.datasets.samples_generator import make_blobs # X為樣本特徵,Y為樣本簇類別, 共1000個樣本,每個樣本3個特徵,共4個簇 X, y = make_blobs(n_samples=10000, n_features=3, centers=[[3,3, 3], [0,0,0], [1,1,1], [2,2,2]], cluster_std=[0.2, 0.1, 0.2, 0.2], random_state =9) fig = plt.figure() ax = Axes3D(fig, rect=[0, 0, 1, 1], elev=30, azim=20) plt.scatter(X[:, 0], X[:, 1], X[:, 2],marker='o')
輸出如圖:
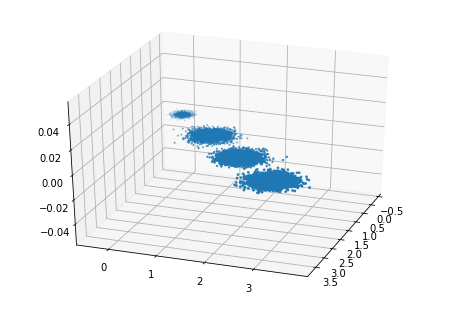
現在我們來進行降維,從3維降到2維,程式碼如下:
from sklearn.decomposition import PCA pca = PCA(n_components=2) pca.fit(X) X_new = pca.transform(X) plt.scatter(X_new[:, 0], X_new[:, 1],marker='o') plt.show()
輸出如圖:

在很多應用中,當我們將資料降維並用於訓練後,訓練出來的模型之後的輸出也是降維後的資料,需要還原回原始維度。這時候需要將pca演算法進行逆運算:
X_old=np.dot(Xnew,pca.components)+pca.mean_
即將新資料和components_相乘並加上平均值。
使用上文的例子,程式碼如下:
X_old=np.dot(X_new,pca.components_)+pca.mean_ fig = plt.figure() ax = Axes3D(fig, rect=[0, 0, 1, 1], elev=30, azim=20) plt.scatter(X_old[:, 0], X_old[:, 1], X_old[:, 2],marker='o')
輸出如圖:

可以看到,資料即是投影到最大方差方向但並未進行降維時的樣子。
*parameter的命名後面沒有下劃線,而attribute的命名後面都有下劃線,以此區分。
