geatpy庫模板moea_nsga2_templet求解多目標問題
阿新 • • 發佈:2018-12-16
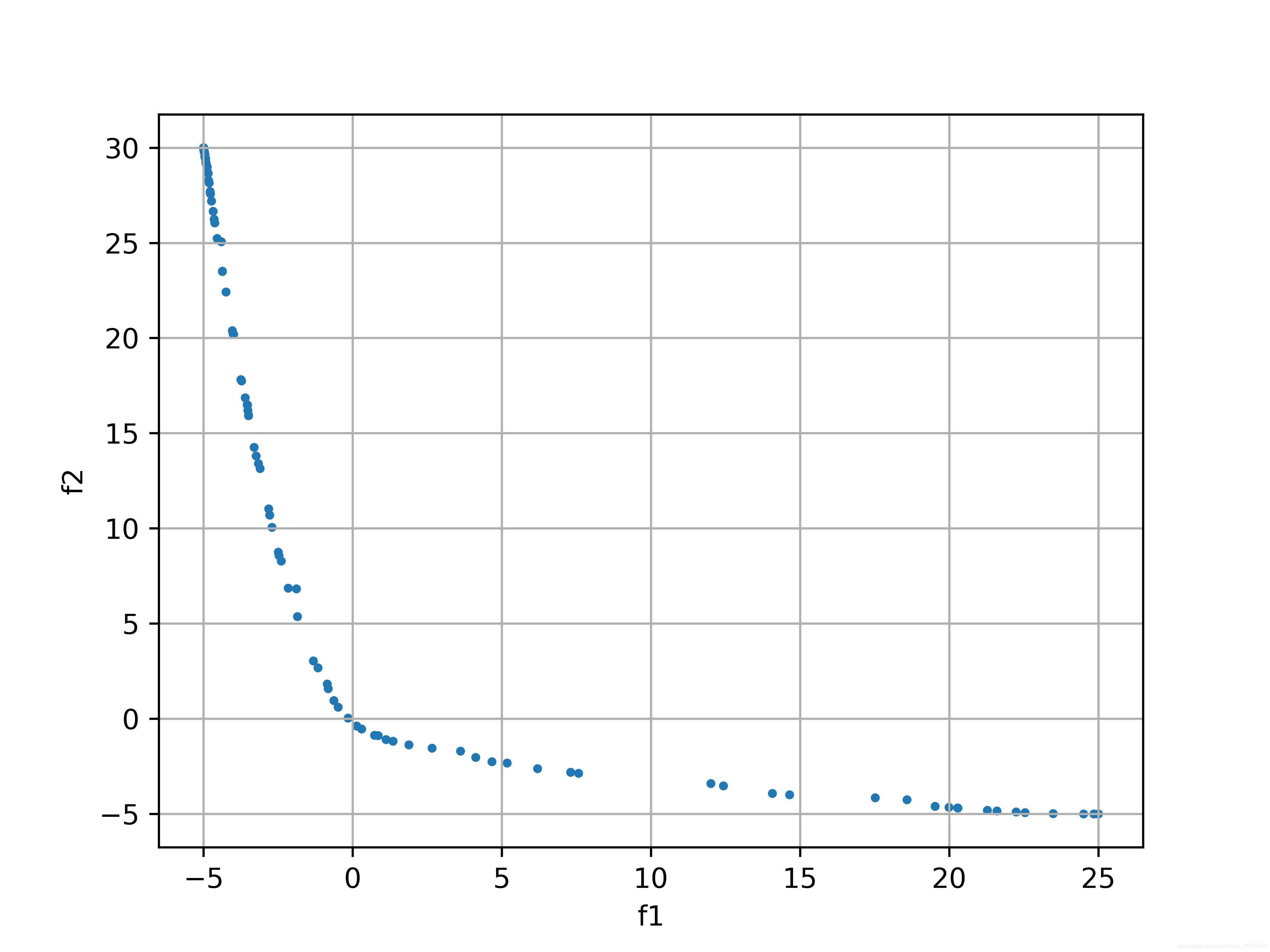
帕累託前沿個數得到的是目標函式值及對應的關係,變數的最優值沒顯示出來。
程式碼(aimfuc.py)
import numpy as np
def aimfuc(x, LegV): # 定義目標函式
x1 = x[:, 0]
x2 = x[:, 1]
fun1 = (x1 - x2) ** 2 + x1
fun2 = x1 ** 2 - x2
return [np.vstack([fun1, fun2]).T, LegV]
程式碼(punishing.py)
# -*- coding: utf-8 -*- import numpy as np def punishing(LegV, FitnV): FitnV[np.where(LegV == 0)[0]] = np.min(FitnV) * 0.1 # 對非可行解嚴厲懲罰 return FitnV
程式碼(main.py)
# -*- coding: utf-8 -*- import geatpy as ga import numpy as np # 獲取函式介面地址 AIM_M = __import__('aimfuc') PUN_M = __import__('punishing') """============================變數設定============================""" x1 = [-5, 5] # 自變數1的範圍 x2 = [-5, 5] # 自變數2的範圍 b1 = [1, 1] # 自變數1是否包含下界 b2 = [1, 1] # 自變數2是否包含上界 precisions = [1, 1] # 在二進位制/格雷碼編碼中代表自變數的編碼精度,當控制變數是二進位制/格雷編碼時,該引數可控制編碼的精度 ranges = np.vstack([x1, x2]).T # 生成自變數的範圍矩陣 borders = np.vstack([b1, b2]).T # 生成自變數的邊界矩陣 # 生成區域描述器 FieldD = ga.crtfld(ranges, borders, precisions) """========================遺傳演算法引數設定=========================""" NIND = 50 # 種群規模 MAXGEN = 500 # 最大遺傳代數 MAXSIZE = 200 # 最大帕累託前沿數 GGAP = 0.8 # 代溝:子代與父代的重複率為(1-GGAP) selectStyle = 'rws' # 遺傳演算法的選擇方式設為"rws"——輪盤賭選擇 recombinStyle = 'xovdp' # 遺傳演算法的重組方式,設為兩點交叉 recopt = 0.9 # 交叉概率 pm = 0.1 # 變異概率 SUBPOP = 1 # 設定種群數為1 maxormin = 1 # 設定標記表明這是最小化目標 """=======================呼叫程式設計模板進行種群進化===================""" # 呼叫程式設計模板進行種群進化,得到種群進化和變數的追蹤器以及執行時間 [ObjV, NDSet, NDSetObjV, times] = ga.moea_nsga2_templet(AIM_M=AIM_M, AIM_F="aimfuc", PUN_M=PUN_M, PUN_F="punishing", FieldDR=FieldD, problem="R", maxormin=1, MAXGEN=MAXGEN, MAXSIZE=MAXSIZE, NIND=NIND, SUBPOP=SUBPOP, GGAP=GGAP, selectStyle=selectStyle, recombinStyle=recombinStyle, recopt=recopt, pm=pm, distribute=True, drawing=1)
測試
用時:39.307276010513306 秒
帕累託前沿點個數:200 個
單位時間找到帕累託前沿點個數:5 個