基於sklearn的分類器實戰
已遷移到我新部落格,閱讀體驗更佳基於sklearn的分類器實戰
完整程式碼實現見github:click me
一、實驗說明
1.1 任務描述

1.2 資料說明
一共有十個資料集,資料集中的資料屬性有全部是離散型的,有全部是連續型的,也有離散與連續混合型的。通過對各個資料集的瀏覽,總結出各個資料集的一些基本資訊如下:
連續型資料集: 1. diabets(4:8d-2c) 2. mozilla4(6:5d-2c) 3. pc1(7:21d-2c) 4. pc5(8:38d-2c) 5. waveform-5000(9:40d-3c) 離散型資料集: 1. breast-w(0:9d-2c-?) 離散-連續混合型資料集: 1. colic(1:22d-2c-?) 2. credit-a(2:15d-2c-?) 3. credit-g(3:20d-2c) 4. hepatitis(少量離散屬性)(5:19d-2c-?)
舉一個例子說明,colic(1:22d-2c-?)對應colic這個資料集,冒號前面的1表示人工標註的資料集序號(在程式碼實現時我是用序號來對映資料集的),22d表示資料集中包含22個屬性,2c表示資料集共有3種類別,'?'表示該資料集中含有缺失值,在對資料處理前需要注意。
二、資料預處理
由於提供的資料集檔案格式是weka的.arff檔案,可以直接匯入到weka中選擇各類演算法模型進行分析,非常簡便。但是我沒有藉助weka而是使用sklearn來對資料集進行分析的,這樣靈活性更大一點。所以首先需要了解.arff的資料組織形式與結構,然後使用numpy讀取到二維陣列中。
具體做法是過濾掉.arff中'%'開頭的註釋,對於'@'開頭的標籤,只關心'@attribute'後面跟著的屬性名與屬性型別,如果屬性型別是以'{}'圍起來的離散型屬性,就將這些離散型屬性對映到0,1,2......,後面讀取到這一列屬性的資料時直接用建好的對映將字串對映到數字。除此之外就是資料內容了,讀完一個數據集的內容之後還需要檢測該資料集中是否包含缺失值,這個使用numpy的布林型索引很容易做到。如果包含缺失值,則統計缺失值這一行所屬類別中所有非缺失資料在缺失屬性上各個值的頻次,然後用出現頻次最高的值來替換缺失值,這就完成對缺失值的填補。具體實現可以參見preprocess.py模組中fill_miss函式。
三、程式碼設計與實現
實驗環境:
python 3.6.7
configparser 3.7.4
scikit-learn 0.20.2
numpy 1.15.4
matplotlib 3.0.3
各個分類器都要用到的幾個模組在這裡做一個簡要說明。
- 交叉驗證: 使用sklearn.model_selection.StratifiedKFold對資料作分層的交叉切分,分類器在多組切分的資料上進行訓練和預測
- AUC效能指標: 使用sklearn.metrics.roc_auc_score計算AUC值,AUC計算對多類(二類以上)資料屬性還需提前轉換成one hot編碼,使用了sklearn,preprocessing.label_binarize來實現,對於多分類問題選擇micro-average
- 資料標準化: 使用sklearn.preprocessing.StandardScaler來對資料進行歸一標準化,實際上就是z分數
3.1 樸素貝葉斯Naive Bayes
由於大部分資料集中都包含連續型屬性,所以選擇sklearn.naive_bayes.GaussianNB來對各個資料集進行處理
clf = GaussianNB()
skf = StratifiedKFold(n_splits=10)
skf_accuracy1 = []
skf_accuracy2 = []
n_classes = np.arange(np.unique(y).size)
for train, test in skf.split(X, y):
clf.fit(X[train], y[train])
skf_accuracy1.append(clf.score(X[test], y[test]))
if n_classes.size < 3:
skf_accuracy2.append(roc_auc_score(y[test], clf.predict_proba(X[test])[:, 1], average='micro'))
else:
ytest_one_hot = label_binarize(y[test], n_classes)
skf_accuracy2.append(roc_auc_score(ytest_one_hot, clf.predict_proba(X[test]), average='micro'))
accuracy1 = np.mean(skf_accuracy1)
accuracy2 = np.mean(skf_accuracy2)在各個資料集上進行交叉驗證後的accuracy和AUC效能指標如下
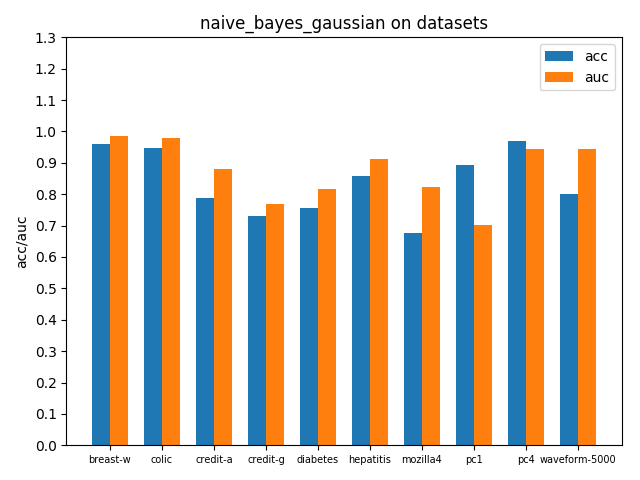
可以看到大部分資料集上的auc指標都比acc高,說明控制好概率閾值(這裡預設0.5)acc可能還有提升空間,因為樣本分佈跟總體分佈還有一定的差距,樣本數布可能很不平衡,並且權衡一個合適的閾值點還需要結合分類問題的背景和關注重點。由於auc指標考慮到了所有可能閾值劃分情況,auc越高能說明模型越理想,總體上能表現得更好。
3.2 決策樹decision tree
使用sklearn.tree.DecisionTreeClassifier作決策樹分析,並且採用gini係數選擇效益最大的屬性進行劃分,下面給出介面呼叫方式,交叉驗證方式與前面的naive bayes一樣
clf = DecisionTreeClassifier(random_state=0, criterion='gini')在各個資料集上進行交叉驗證後的accuracy和AUC效能指標如下
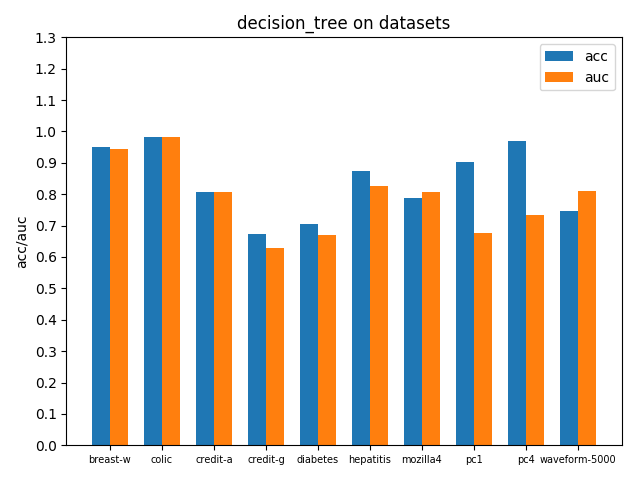
3.3 K近鄰KNN
使用sklearn.neighbors.KNeighborsClassifier實現KNN,鄰居數設定為3即根據最近的3個鄰居來投票抉擇樣本所屬分類,因為資料集最多不超過3類,鄰居數選擇3~6較為合適,設得更高效果增益不明顯並且時間開銷大,下面給出介面呼叫方式
clf = KNeighborsClassifier(n_neighbors=3)在各個資料集上進行交叉驗證後的accuracy和AUC效能指標如下
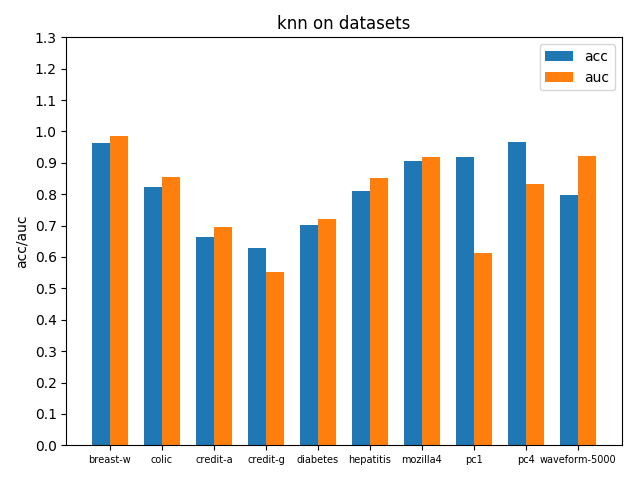
3.4 神經網路之多層感知機MLP
使用sklearn.neural_network.MLPClassifier實現MLP,設定一層隱藏層100個節點,啟用函式relu,優化器Adam,學習率1e-3並且自適應降低,l2正則項懲罰係數,下面給出具體的介面呼叫方式以及引數配置
clf = MLPClassifier(hidden_layer_sizes=(100),
activation='relu',
solver='adam',
batch_size=128,
alpha=1e-4,
learning_rate_init=1e-3,
learning_rate='adaptive',
tol=1e-4,
max_iter=200)在各個資料集上進行交叉驗證後的accuracy和AUC效能指標如下
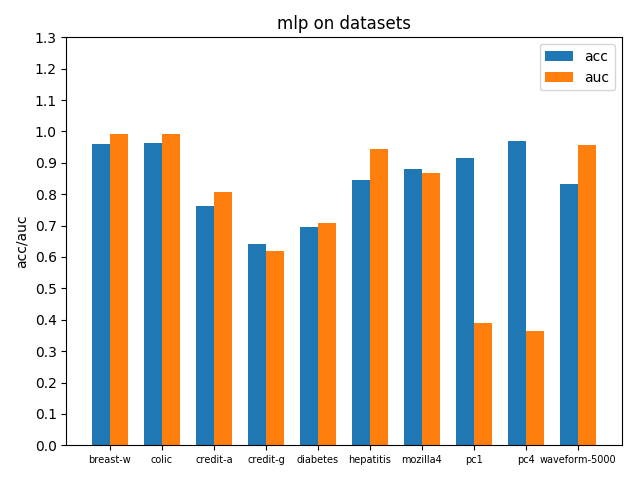
3.5 支援向量機SVM
使用sklean.svm.LinearSVC實現線性SVM分類器,介面呼叫方式如下
clf = LinearSVC(penalty='l2', random_state=0, tol=1e-4)在各個資料集上進行交叉驗證後的accuracy和AUC效能指標如下
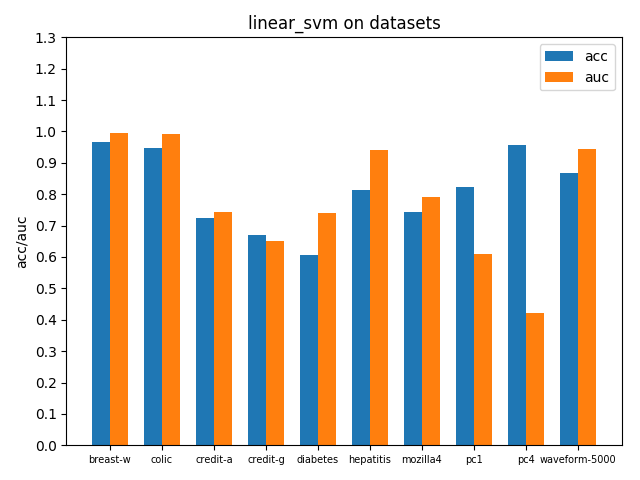
四、實驗結果與分析
4.1 不同資料集上的模型對比
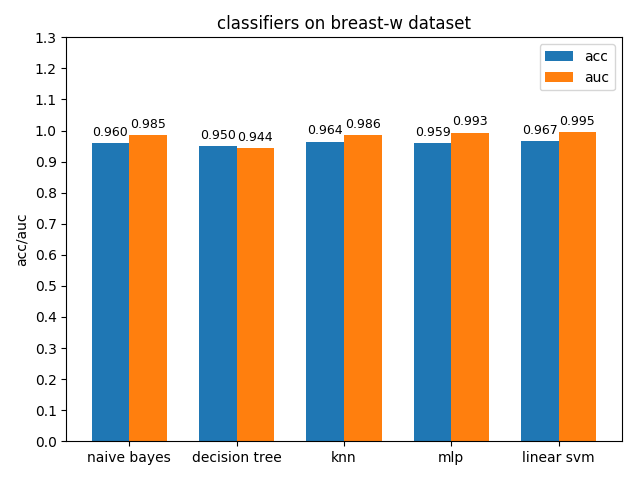
4.1.1 breast-w dataset
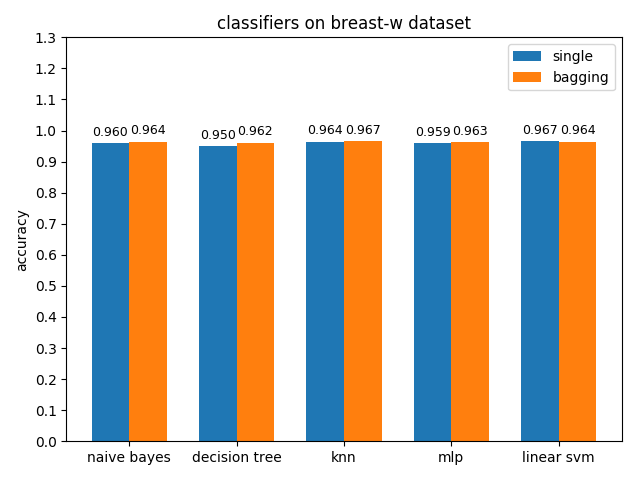
breast-w資料集上,各分類模型的效果都很好,其中linear svm的準確率最高,mlp的auc值最高
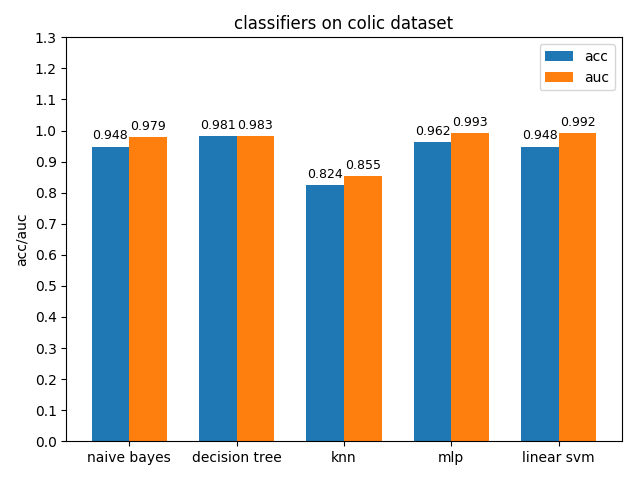
4.1.2 colic dataset
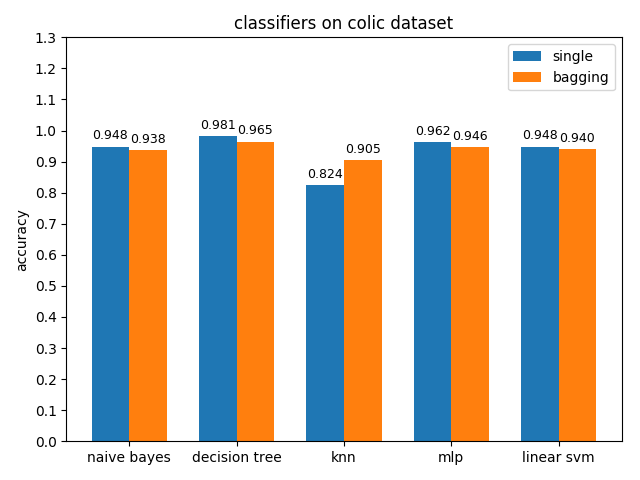
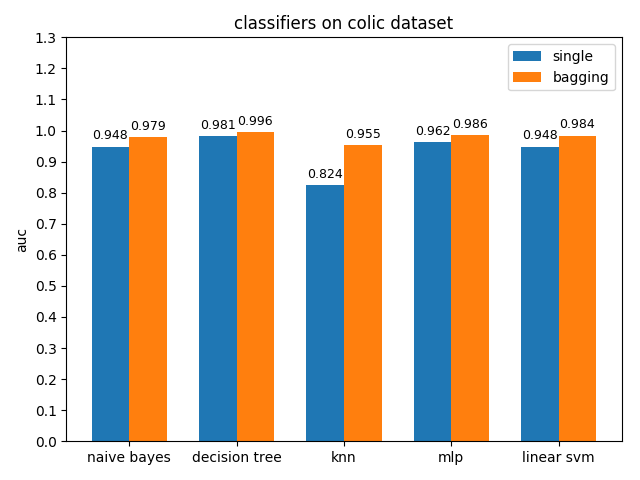
colic資料集上,knn效果不佳,其它分類模型的效果都很好,其中decision tree的準確率最高,mlp的auc值最高
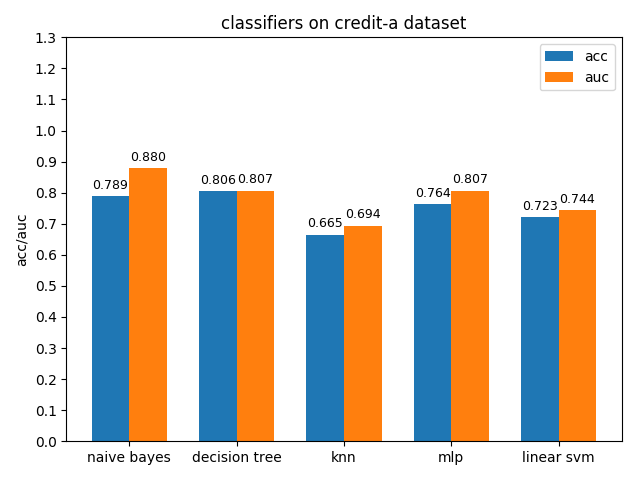
4.1.3 credit-a dataset
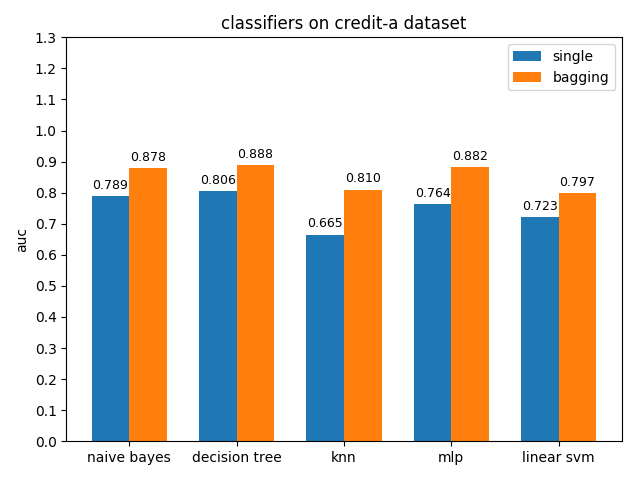
credit-a資料集上,各分類模型的效果都不是很好,其中decision tree的準確率最高,naive bayes的auc值最高
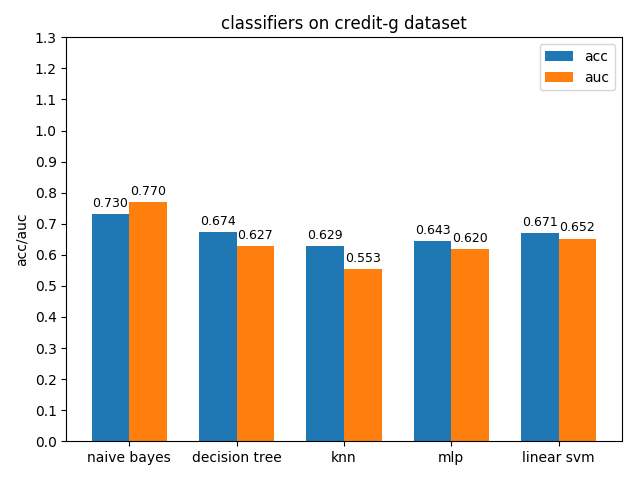
4.1.4 credit-g dataset
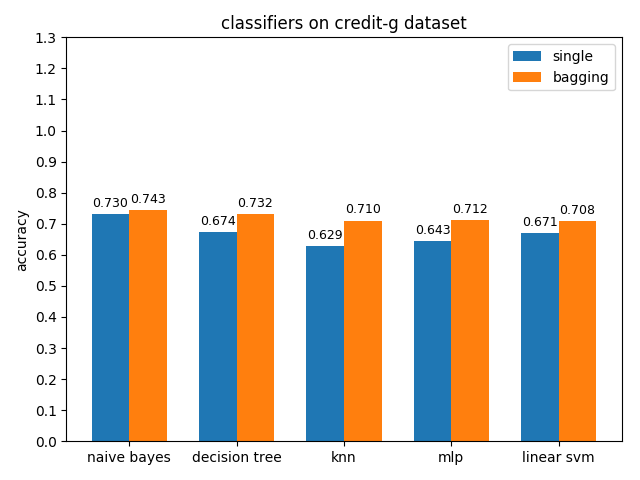
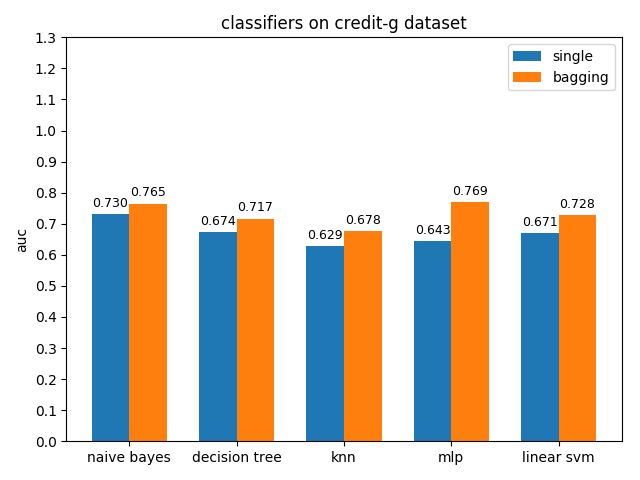
credit-a資料集上,各分類模型的效果都不是很好,其中naive bayes的準確率和auc值都是最高的
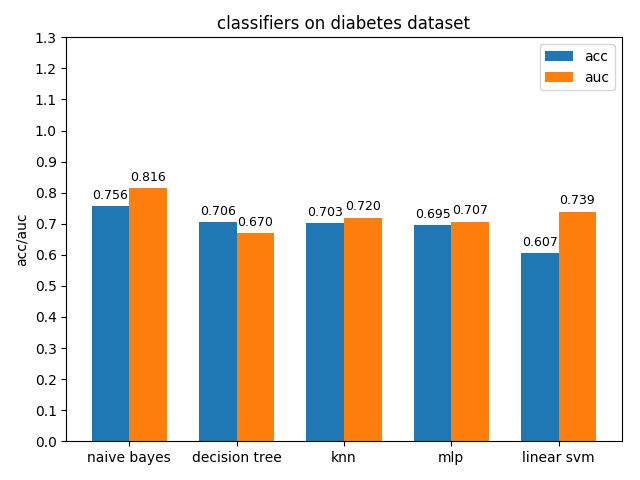
4.1.5 diabetes dataset
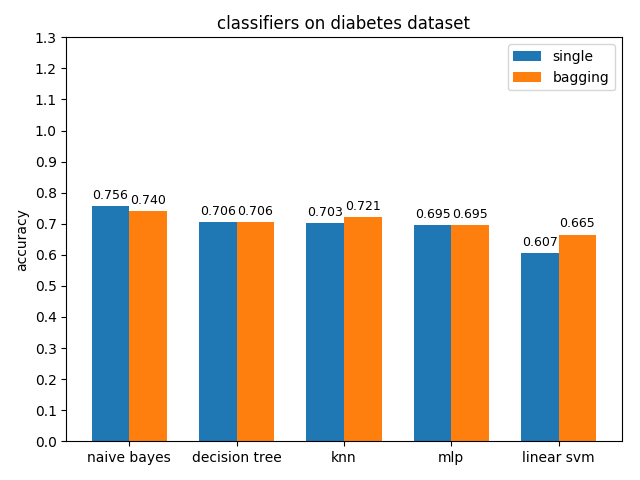
diabetes資料集上,各分類模型的效果都不是很好,其中naive bayes的準確率和auc值都是最高的
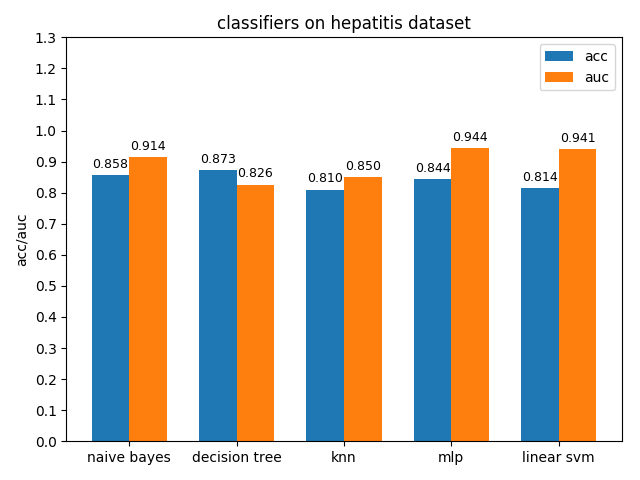
4.1.6 hepatitis dataset
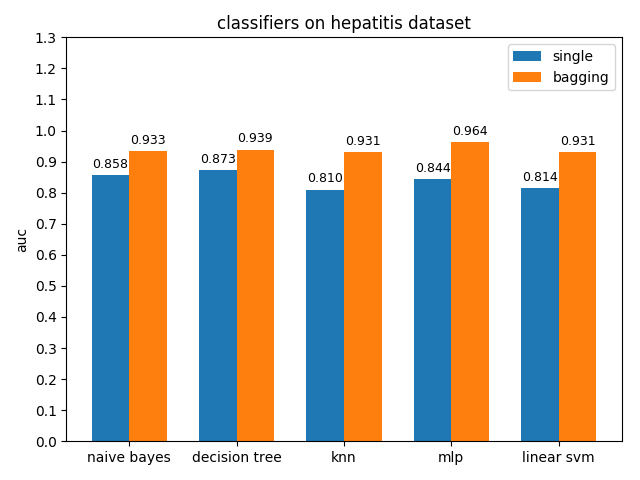
hepatitis資料集上,各分類模型的準確率都沒達到90%,decision tree的準確率最高,mlp的auc值最高,但是各分類模型的auc值基本都比acc高除了decision tree,說明hepatitis資料集的資料分佈可能不太平衡
通過weka對hepatitis資料集上的正負類進行統計得到下面的直方圖

從上面的直方圖可以驗證之前的猜測是對的,hepatitis資料集正負類1:4,資料分佈不平衡,正類遠少於負類樣本數
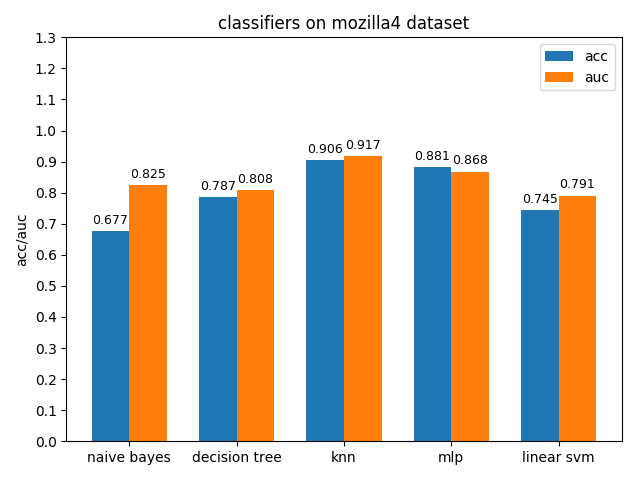
4.1.7 mozilla4 dataset
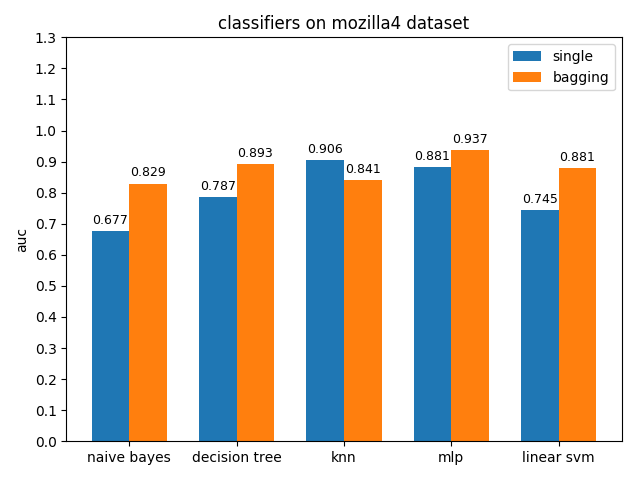
mozilla4資料集上,各分類模型的表現差異很大,其中knn的acc和auc都是最高的,naivie bayes的acc和auc相差甚大
4.1.8 pc1 dataset
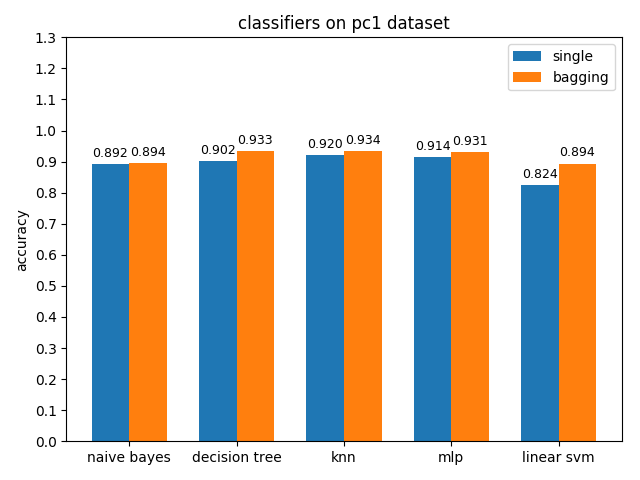
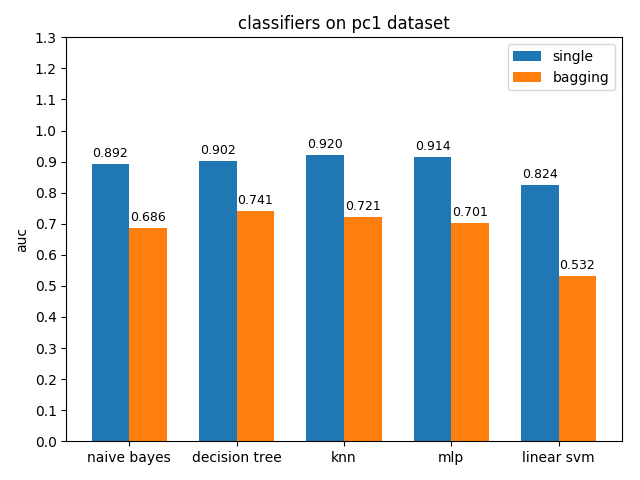
pc1資料集上,各分類模型的準確率基本都挺高的,但是auc值普遍都很低,使用weka對資料進行統計分析後發現pc1資料集的正負類比達到13:1,根據auc計算原理可知正類太多可能會導致TPR相比FPR會低很多,從而壓低了auc值
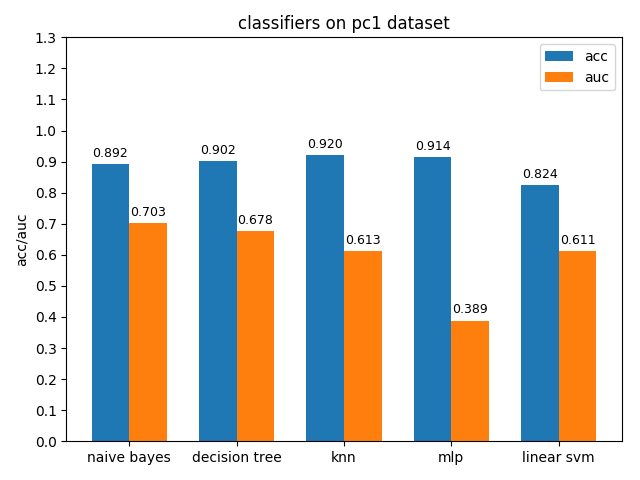
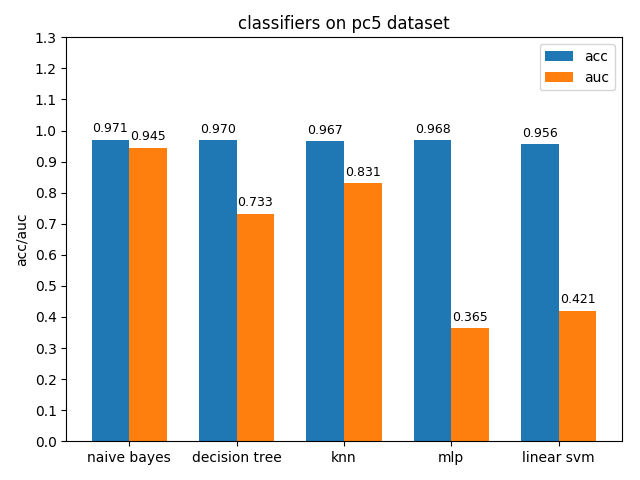
4.1.9 pc5 dataset
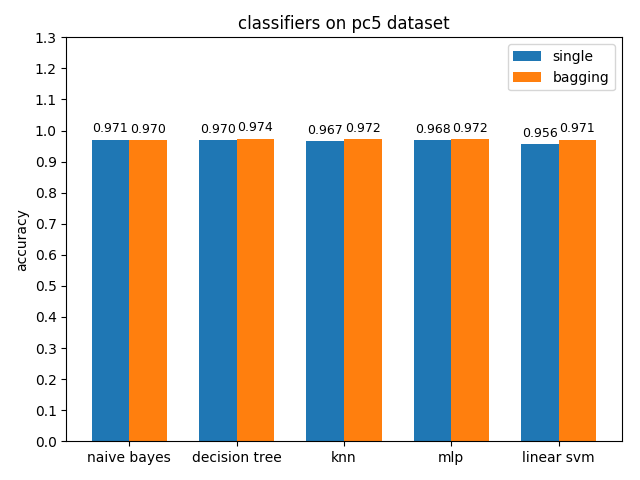
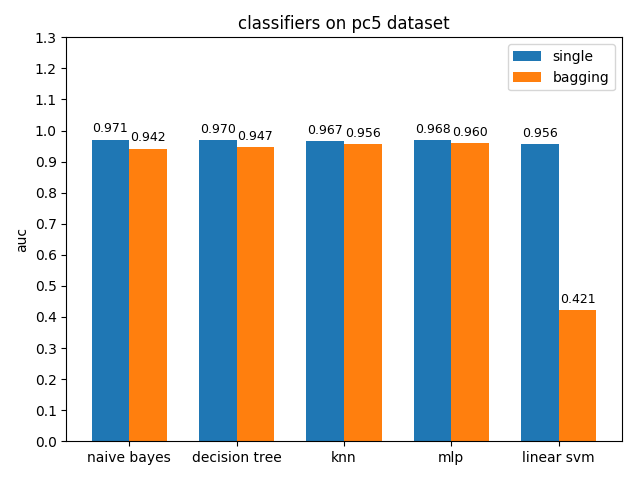
pc5資料集上,各分類模型的準確率都達到了90%以上,但是auc都比acc要低,其中mlp和linear svm的acc與auc相差甚大,原因估計和pc1差不多,正類樣本太多拉低了AUC,使用weka分析後發現pc5正負類樣本比值達到了32:1,並且資料中夾雜著些許異常的噪聲點
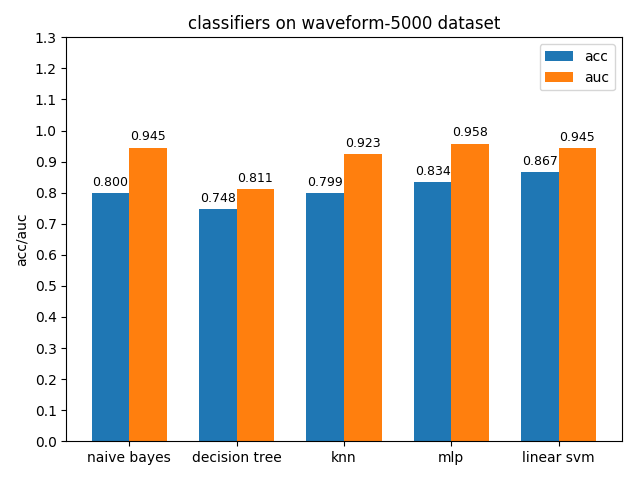
4.1.10 waveform-5000 dataset
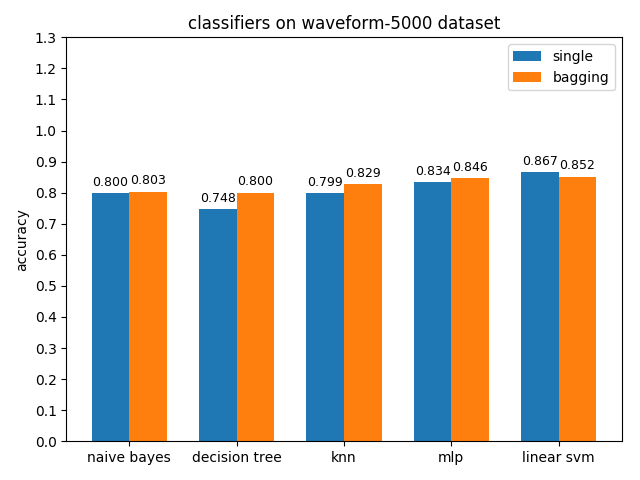
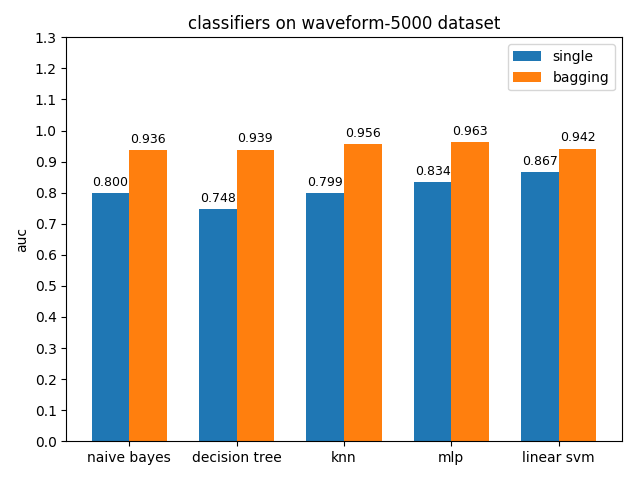
waveform-5000資料集上,各分類模型的準確率基本都是在80%左右,各分類模型的auc基本都有90%除了decision tree以外。waveform-5000是一個三類別的資料集,相比前面的2分類資料集預測難度也會更大,概率閾值的選擇尤為關鍵,一個好的閾值劃分會帶來更高的準確率。
4.2 模型的bagging和single效能對比
4.2.1 breast-w dataset
準確率對比
AUC對比
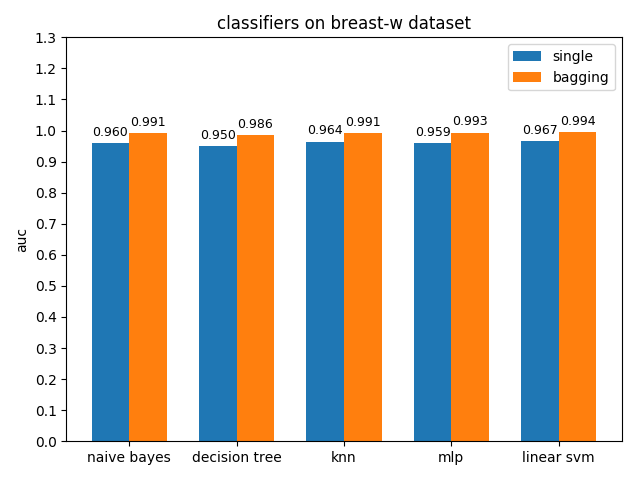
4.2.1 colic dataset
準確率對比
AUC對比
4.2.3 credit-a dataset
準確率對比
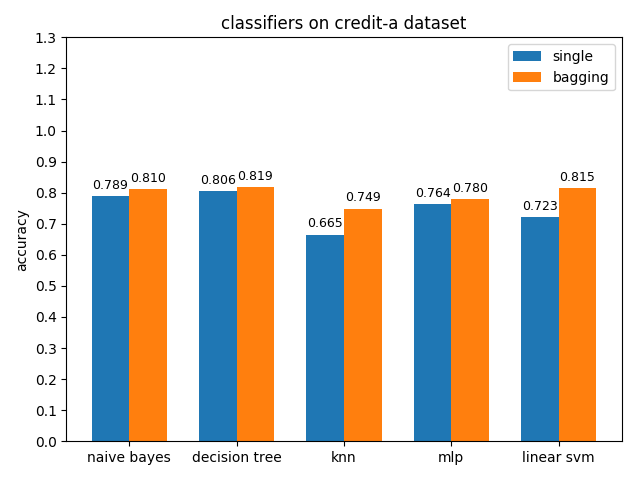
AUC對比
4.2.4 credit-g dataset
準確率對比
AUC對比
4.2.5 diabetes dataset
準確率對比
AUC對比
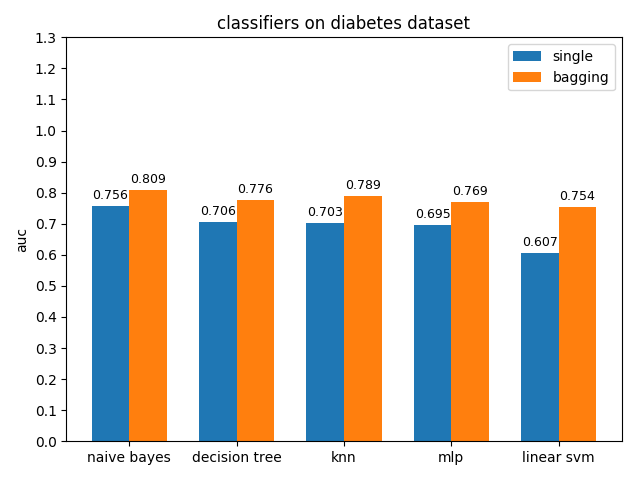
4.2.6 hepatitis dataset
準確率對比
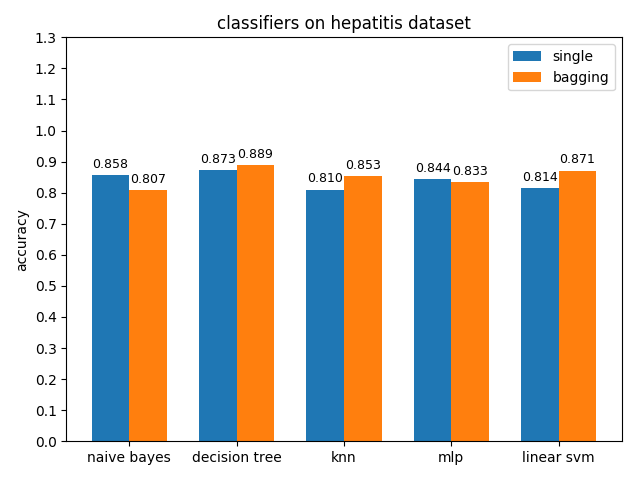
AUC對比
4.2.7 mozilla4 dataset
準確率對比
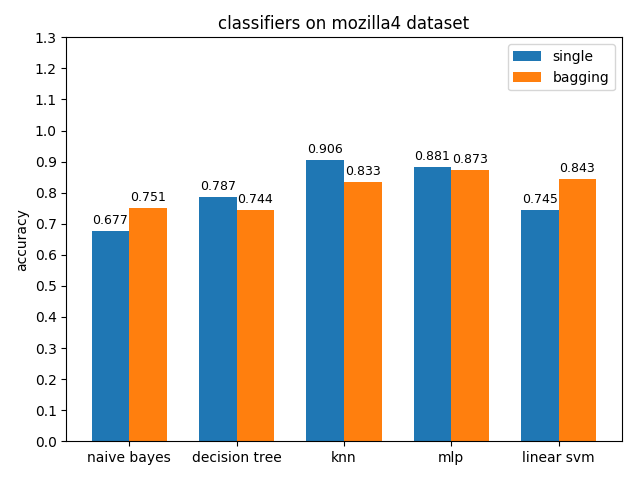
AUC對比
4.2.8 pc1 dataset
準確率對比
AUC對比
4.2.9 pc5 dataset
準確率對比
AUC對比
4.2.10 waveform-5000 dataset
準確率對比
AUC對比
五、優化
5.1 降維
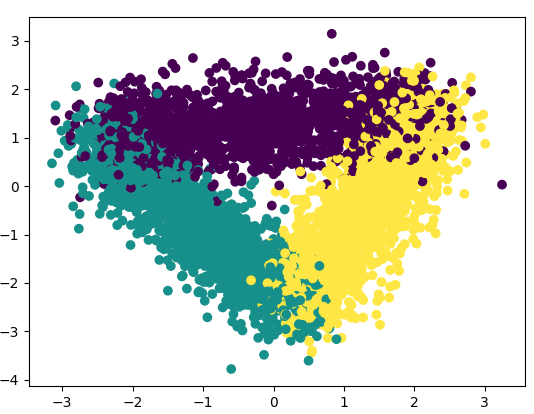
pc1,pc5,waveform-5000,colic,credit-g這幾個資料集的屬性維度都在20維及以上,而對分辨樣本類別有關鍵作用的就只有幾個屬性,所以想到通過降維來擯除干擾屬性以使分類模型更好得學習到資料的特徵。用主成分分析(PCA)降維之後的資料分類效果不佳,考慮到都是帶標籤的資料,就嘗試使用線性判別分析(LDA)利用資料類別資訊來降維。但是LDA最多降維到類別數-1,對於二類資料集效果不好。waveform-5000包含三種類別,於是就嘗試用LDA對waveform-5000降維之後再使用各分類模型對其進行學習。
使用sklearn.discriminant_analysis.LinearDiscriminantAnalysis對waveform-5000降維之後的資料樣本分佈散點圖如下,可以明顯看到資料被聚為三類,降維之後的資料特徵資訊更為明顯,干擾資訊更少,對分類更有利
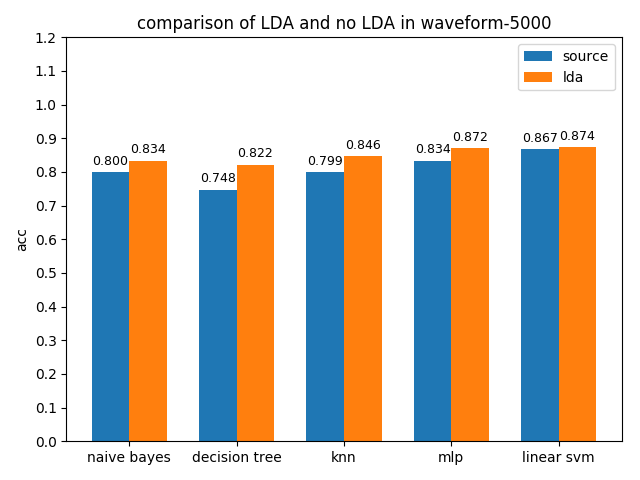
各分類模型在原資料集和LDA降維資料集上準確率對比如下圖
各分類模型在原資料集和LDA降維資料集上AUC值對比如下圖
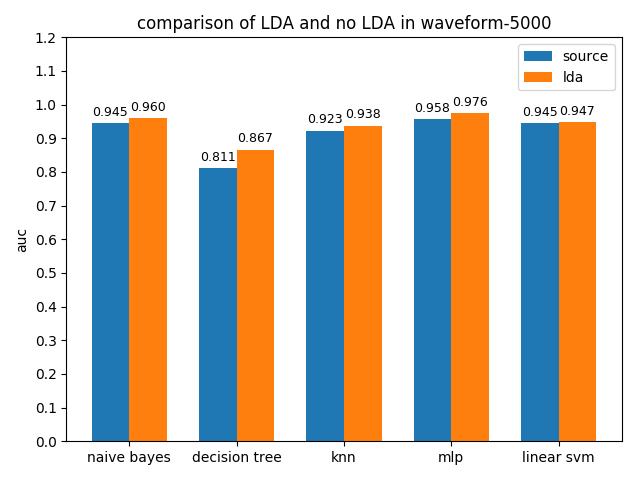
可以看到降維之後的分類效果很理想,無論是acc還是auc,各個分類模型都得到了不同程度的效能提升
5.2 標準化提升bagging with KNN效能
由於KNN是基於樣本空間的歐氏距離來計算的,而多屬性樣本的各屬性通常有不同的量綱和量綱單位,這無疑會給計算樣本之間的真實距離帶來干擾,影響KNN分類效果。為了消除各屬性之間的量綱差異,需要進行資料標準化處理,計算屬性的z分數來替換原屬性值。在具體的程式設計時使用sklearn.preprocessing.StandardScaler來對資料進行標準化。
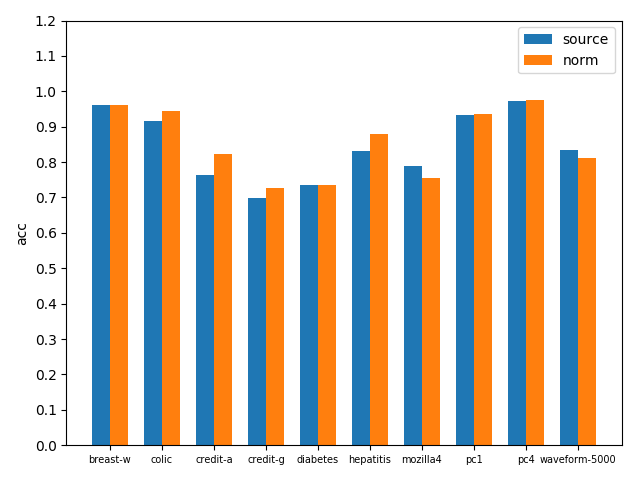
bagging with KNN在原資料集和標準化資料集上準確率對比如下圖
bagging with KNN在原資料集和標準化資料集上AUC對比如下圖
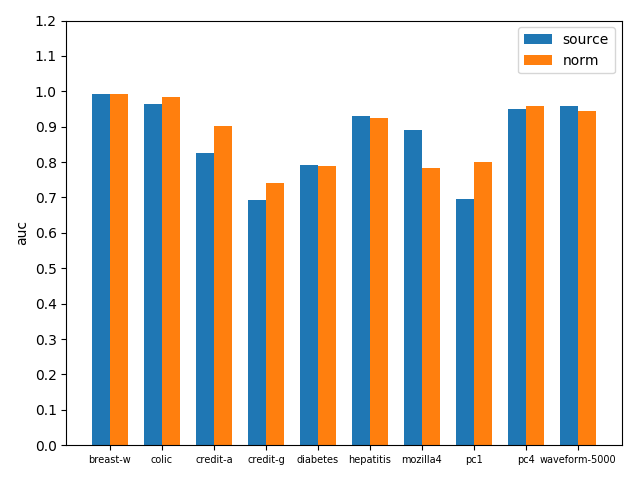
可以看到標準化之後效果還是不錯的,無論是acc還是auc,基本在各資料集上都得到了效能提升
六、實驗總結
此次實驗讓我對各個分類器的具體用法有了初步的瞭解,不同的資料集上應用不同分類模型的效果截然不同。資料探勘任務70%時間消耗在資料清洗上,無論什麼分類模型,其在高質量資料上的表現肯定是要優於其在低質量資料上的表現。所以拿到一個數據集後不能貿然往上面套分類模型,而是應該先對資料集進行觀察分析,然後利用工具對資料進行清洗、約簡、整合以及轉換,提升資料質量,讓分類更好得學習到資料的特徵,從而有更好的分類效果。本次實驗我也對各資料集進行了預處理,包括資料缺失值填補、資料型別轉換、資料降維、資料標準化等等,這些工作都在各分類模型的分類效果上得到了一定的反饋。實驗過程中也遇到了一些問題,比如使用sklearn.metrics.roc_auc_score計算多類別資料的AUC時需要提前將資料標籤轉換為one hot編碼,LDA最多隻能將資料維度降維到類別數-1等等,這些都為我以後進行資料探勘工作積累了寶貴經驗
