FPKM\RPKM\TPM學習[轉載]
轉自:http://www.360doc.com/content/18/0112/02/50153987_721216719.shtml
1.問題提出
在RNA-Seq的分析中,對基因或轉錄本的read counts數目進行標準化(normalization)是一個極其重要的步驟,因為落在一個基因區域內的read counts數目取決於基因長度和測序深度。
很容易理解,一個基因越長,測序深度越高,落在其內部的read counts數目就會相對越多。
當我們進行基因差異表達的分析時,往往是在多個樣本中比較不同基因的表達量,如果不進行資料標準化,比較結果是沒有意義的。
因此,我們需要標準化的兩個關鍵因素就是基因長度和測序深度,常常用RPKM (Reads Per Kilobase Million), FPKM (Fragments Per Kilobase Million) 和 TPM (Transcripts Per Million)作為標準化數值。
那麼,這三者計算原理是什麼,有何區別呢?
2.例子說明
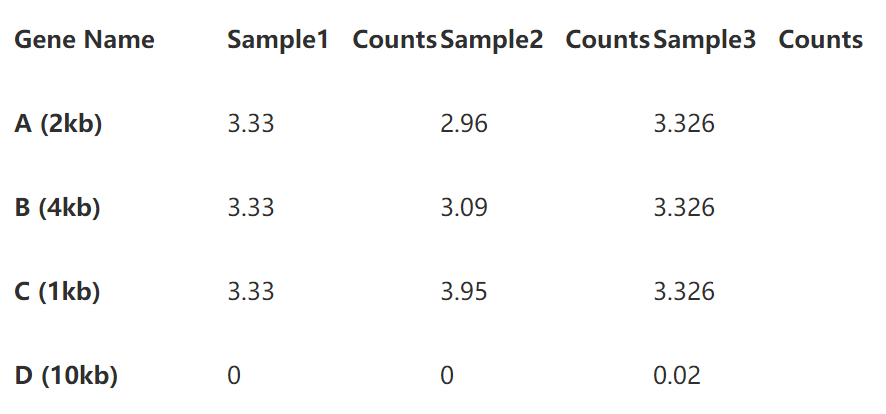
為了更清楚的展示計算過程,我們用三個樣本的4個基因的read counts矩陣做例子。
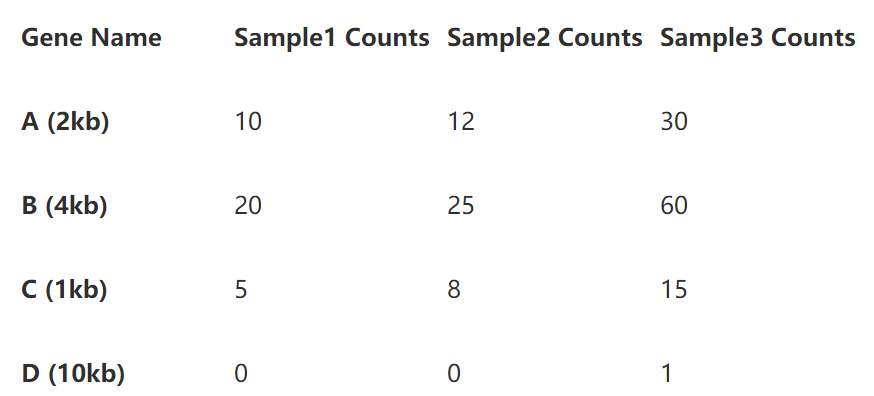
從上表,樣本3的4個基因read counts數目明顯多於其他兩個樣本,說明其測序深度較高,基因B的長度的基因A的兩倍,也使得其read counts在三個樣本中都高於A。
接下來我們要做就是對這個矩陣進行標準化,分別計算RPKM, FPKM和TPM, 請睜大你的眼睛(為了使數值可讀性更好,下面的計算中我們用10代表million)。
2.1計算RPKM
第一步先將測序深度標準化,計算方法很簡單,先分別計算出每個樣本的總reads數(這裡以10為單位),然後將表中資料分別除以總reads數即可,這樣就得到了reads per million. 如下表:
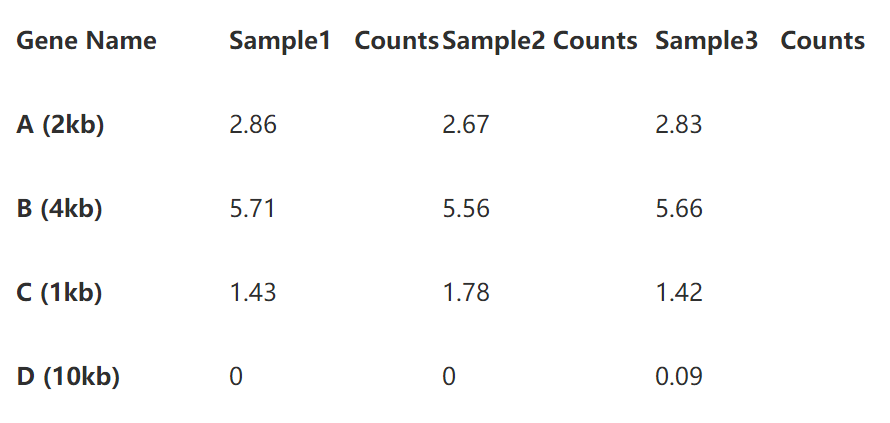
第二步即是基因長度的標準化了。將表2的read per million直接除以基因長度即可,如下表:
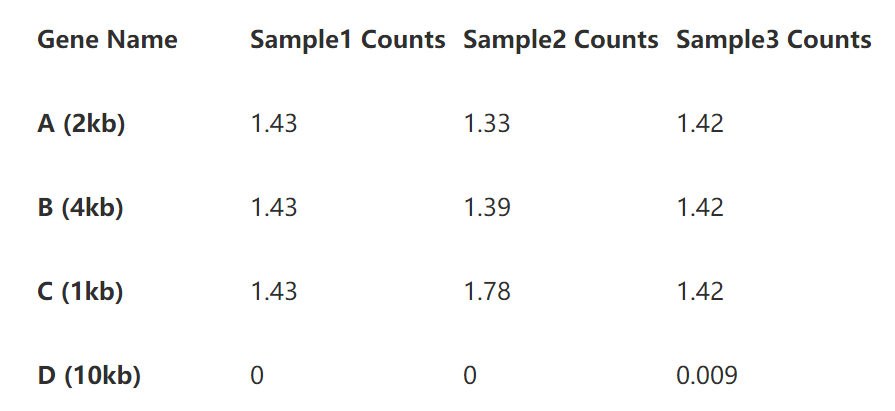
到這裡,我們即得到了RPKM。
2.2FPKM
FPKM和RPKM的定義是相同的,唯一的區別是FPKM適用於雙端測序文庫,而RPKM適用於單端測序文庫。
FPKM會將配對比對到一個片段(fragment)上的兩個reads計算一次,接下來的計算過程跟RPKM一樣。
在兩種測序中


在FPKM中,F就是指fragment,也就是說在計算時使用的是fragment而不是reads。
那麼由此可以得到在雙端測序中, 如果使用RPKM計數(計數reads數目)的話,那麼RPKM值將會是FPKM值的1~2倍。(呼!我終於理解了!)
2.3TPM
終於輪到TPM登場了。
雖然同樣是標準化測序深度和基因長度,TPM的不同在於它的處理順序是不同的。
即先考慮基因長度,再是測序深度。我們仍以表1的那個例子來說明TPM是計算過程。
第一步直接除以基因長度,得到reads per kilobase,如下表:
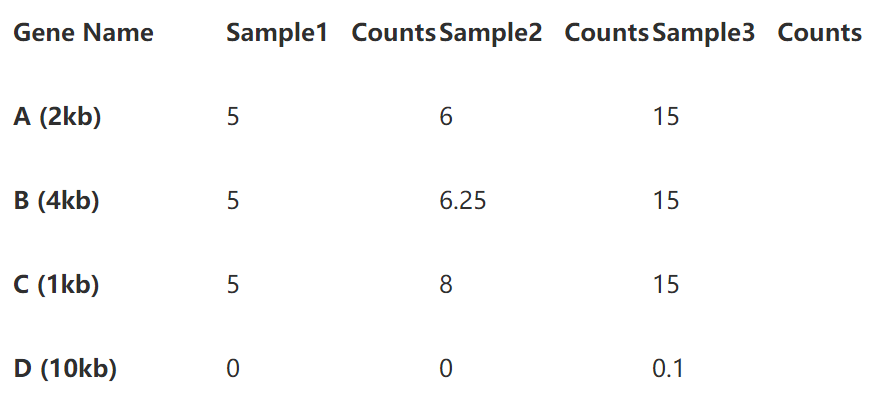
第二步標準化測序深度時,總的reads數要用第一步中除過基因長度的數值。
即第一樣本除以15,第二個樣本除以20.25,第三個樣本除以45.1 (別忘了我們的單位是10哦)。下表就是你們想要的TPM了。
3.RPKM與TPM比較
下面,是考驗你們數學功底的時候了,有沒有看出來TPM分分鐘完虐FPKM/RPKM?其實,只要我們在表3和表5下面多加一行你就能很輕鬆地看到區別了。
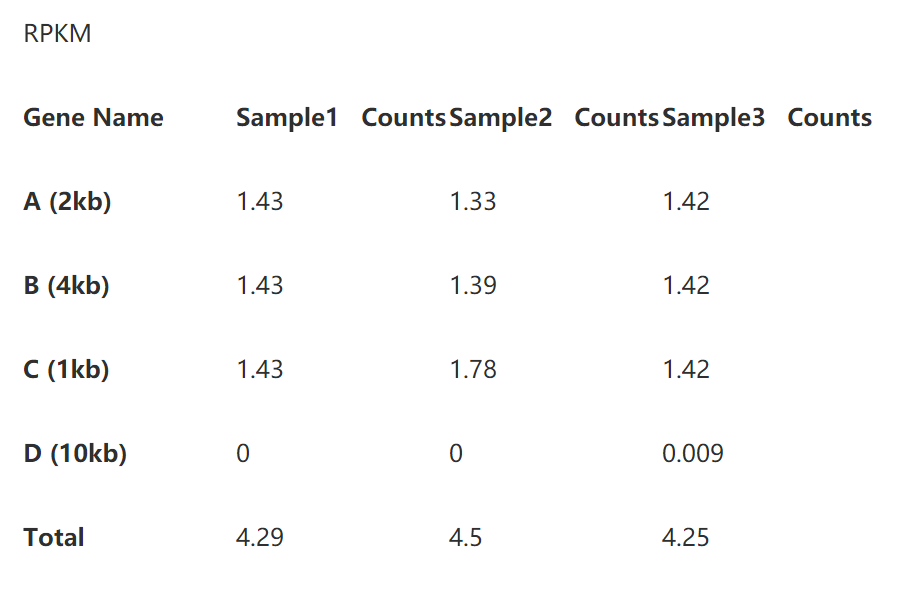
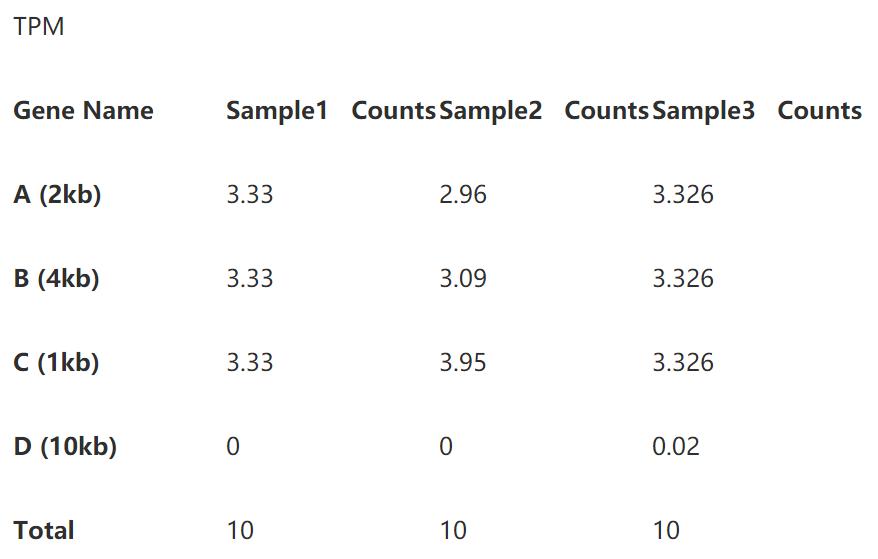
我們看到每個樣本的TPM的總和是相同的,這就意味著TPM數值能體現出比對上某個基因的reads的比例,使得該數值可以直接進行樣本間的比較。
//至於為什麼每個sample的和是相通的,我也沒有搞清楚。
